20 Решения заданий
You can omit tasks with asterisks (*): these are the tasks of higher difficulty, so you need to think a lot on them, not just apply tools you’ve just learned.
20.1 Intro to R
- Divide 9801 by 9.
9801/9## [1] 1089- Calculate logarithm of 2176782336 with base 6.
log(2176782336, 6)## [1] 12- Calculate natural logarithm of 10 and multiply it by 5.
log(10)*5## [1] 11.51293- Using function
sin()calculate \(\sin (\pi), \sin \left(\frac{\pi}{2}\right), \sin \left(\frac{\pi}{6}\right)\).
The value of \(\pi\) is a predefined constant in R (
pi).
sin(pi)## [1] 1.224647e-16sin(pi/2)## [1] 1sin(pi/6)## [1] 0.520.2 Vectors creation
- Create a vector with values 2, 30 and 4000.
c(2, 30, 4000)## [1] 2 30 4000- Create a vector from 1 to 20.
1:20## [1] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20- Create a vector from 20 to 1.
20:1## [1] 20 19 18 17 16 15 14 13 12 11 10 9 8 7 6 5 4 3 2 1- Function
sum()returns sum of all elements in input. Calculate sum of the first 100 natural numbers (i.e. all whole numbers from 1 to 100).
sum(1:100)## [1] 5050- Create a vector from 1 to 20 and then back to 1. Number 20 must appear only once!
c(1:20, 19:1)## [1] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 19 18 17 16 15
## [26] 14 13 12 11 10 9 8 7 6 5 4 3 2 1- Create a vector with values 5, 4, 3, 2, 2, 3, 4, 5:
c(5:2, 2:5)## [1] 5 4 3 2 2 3 4 5- Create a vector 2, 4, 6, … , 18, 20.
seq(2, 20, 2)## [1] 2 4 6 8 10 12 14 16 18 20- Create a vector 0.1, 0.2, 0.3, …, 0.9, 1.
seq(0.1, 1, 0.1)## [1] 0.1 0.2 0.3 0.4 0.5 0.6 0.7 0.8 0.9 1.0- 2020 year is a leap year. The next leap year will be 4 years later — it will be year 2024. Create a calendar of all leap years for the XXI century since year 2020.
2100 year belongs to the XXI century, not the XXII.
seq(2020, 2100, 4)## [1] 2020 2024 2028 2032 2036 2040 2044 2048 2052 2056 2060 2064 2068 2072 2076
## [16] 2080 2084 2088 2092 2096 2100- Create a vector containing 20 repetitions of “Hey!”
rep("Hey!", 20)## [1] "Hey!" "Hey!" "Hey!" "Hey!" "Hey!" "Hey!" "Hey!" "Hey!" "Hey!" "Hey!"
## [11] "Hey!" "Hey!" "Hey!" "Hey!" "Hey!" "Hey!" "Hey!" "Hey!" "Hey!" "Hey!"- As I said, many functions that work with scalars (one value), also work well with vectors that contain more than one value. Try to calculate square root of numbers from 1 to 10 using function
sqrt()and svae the result in a variableroots.
roots <- sqrt(1:10)
roots## [1] 1.000000 1.414214 1.732051 2.000000 2.236068 2.449490 2.645751 2.828427
## [9] 3.000000 3.162278- Let’s check that it is really square roots by calculating the 2nd degree of all values of
roots!
roots ^ 2## [1] 1 2 3 4 5 6 7 8 9 10- If everything is correct you can get the same result by multiplying the
rootsby itself.
roots * roots## [1] 1 2 3 4 5 6 7 8 9 10- Create a vector with one 1, two 2, three 3, … , nine 9.
rep(1:9, 1:9)## [1] 1 2 2 3 3 3 4 4 4 4 5 5 5 5 5 6 6 6 6 6 6 7 7 7 7 7 7 7 8 8 8 8 8 8 8 8 9 9
## [39] 9 9 9 9 9 9 920.3 Coercion
- Create a vector
vec1that combines3and values"My"and"vector".
vec1 <- c(3, "My", "vector")
vec1## [1] "3" "My" "vector"- Try to substitute
TRUEfrom 10. What happens and why?
10 - TRUE## [1] 9- Combine values
10andTRUEin a vectorvec2.
vec2 <- c(10, TRUE)
vec2## [1] 10 1- Combine vector
vec2and value"r":
c(vec2, "r")## [1] "10" "1" "r"- Combine values
10,TRUE,"r"to a vector.
c(10, TRUE, "r")## [1] "10" "TRUE" "r"20.4 Vectorisation
- Create a vector
pwith values 4, 5, 6, 7, and vectorq, with values 0, 1, 2, 3.
p <- 4:7
p## [1] 4 5 6 7q <- 0:3
q## [1] 0 1 2 3- Calculate element-wise sum of
pandq:
p + q## [1] 4 6 8 10- Calculate element-wise difference of
pandq:
p - q## [1] 4 4 4 4- Divide all values of
pby corresponding value ofq:
Yep, you will divide by 0!
p / q## [1] Inf 5.000000 3.000000 2.333333- Raise each value of
pto the corresponding power fromq.
p ^ q## [1] 1 5 36 343- Multiply each value of
pby 10
p * 10## [1] 40 50 60 70- Create a vector of squares for numbers from 1 to 10:
(1:10)^2## [1] 1 4 9 16 25 36 49 64 81 100- Create a vector with values 0, 2, 0, 4, … , 18, 0, 20.
1:20 * 0:1## [1] 0 2 0 4 0 6 0 8 0 10 0 12 0 14 0 16 0 18 0 20- Create a vector with values 1, 0, 3, 0, 5, …, 17, 0, 19, 0.
1:20 * 1:0## [1] 1 0 3 0 5 0 7 0 9 0 11 0 13 0 15 0 17 0 19 0- Create a vector with first 20 powers of 2.
2 ^ (1:20)## [1] 2 4 8 16 32 64 128 256 512
## [10] 1024 2048 4096 8192 16384 32768 65536 131072 262144
## [19] 524288 1048576- Create a vector with values 1, 10, 100, 1000, 10000:
10 ^ (0:4)## [1] 1 10 100 1000 10000- Calculate a sum of a sequence \(\frac{1}{1 \cdot 2}+\frac{1}{2 \cdot 3}+\frac{1}{3 \cdot 4}+\ldots+\frac{1}{50 \cdot 51}\).
sum(1 / (1:50 * 2:51))## [1] 0.9803922- Calculate a sum of a sequence \(\frac{1}{2^{0}}+\frac{1}{2^{1}}+\frac{1}{2^{2}}+\frac{1}{2^{3}}+\ldots \frac{1}{2^{20}}\).
sum(1 / 2 ^ (0:20))## [1] 1.999999- Calculate a sum of a sequence \(1+\frac{4}{3}+\frac{7}{9}+\frac{10}{27}+\frac{13}{81}+\ldots+\frac{28}{19683}\).
sum((3 * (1:10) - 2) / 3 ^ (0:9))## [1] 3.749174- How many numbers from sequence \(1+\frac{4}{3}+\frac{7}{9}+\frac{10}{27}+\frac{13}{81}+\ldots+\frac{28}{19683}\) are bigger than 0.5?
sum((3 * (1:10) - 2) / 3 ^ (0:9) > 0.5)## [1] 320.5 Indexing vectors
- Create a vector
troikiwith values 3, 6, 9, …, 24, 27.
troiki <- seq(3, 27, 3)
troiki## [1] 3 6 9 12 15 18 21 24 27- Extract the 2nd, 5th and 7th value from the vector
troiki.
troiki[c(2, 5, 7)]## [1] 6 15 21- Extract the last but one value from the vector
troiki.
troiki[length(troiki) - 1]## [1] 24- Exctract all values except the last but one from the vector
troiki:
troiki[-(length(troiki) - 1)]## [1] 3 6 9 12 15 18 21 27- Create a vector
vec3:
vec3 <- c(3, 5, 2, 1, 8, 4, 9, 10, 3, 15, 1, 11)- Find the second value of the vector
vec3.
vec3[2]## [1] 5- Find the 2nd and the 5th value from the vector
vec3.
vec3[c(2, 5)]## [1] 5 8- Try to extract the 100th value of the vector
vec3:
vec3[100]## [1] NA- Return all values from the vector
vec3except the second one.
vec3[-2]## [1] 3 2 1 8 4 9 10 3 15 1 11- Return all values from the vector
vec3except the second and the fifth values.
vec3[c(-2, -5)]## [1] 3 2 1 4 9 10 3 15 1 11- Find the last value of the vector
vec3.
vec3[length(vec3)]## [1] 11- Return all the values of the vector
vec3except the first one and the last one.
vec3[c(-1, -length(vec3))]## [1] 5 2 1 8 4 9 10 3 15 1- Find all values of the vector
vec3, that are more than 4.
vec3[vec3 > 4]## [1] 5 8 9 10 15 11- Find all values of the vector
vec3, that are more than 4 but less than 10.
Use logical operators if you want to do the task in one line of code!
vec3[vec3 > 4 & vec3 < 10]## [1] 5 8 9- Find all values of the vector
vec3, that are less than 4 or more than 10.
vec3[vec3 < 4 | vec3 > 10]## [1] 3 2 1 3 15 1 11- Return squares for each value of the vector
vec3.
vec3 ^ 2## [1] 9 25 4 1 64 16 81 100 9 225 1 121- *Calculate square for each value in odd position and calculate square root for each value in even position of the vector
vec3.
Calculating square root is identical to calculating of power of 0.5.
vec3 ^ c(2, 0.5)## [1] 9.000000 2.236068 4.000000 1.000000 64.000000 2.000000 81.000000
## [8] 3.162278 9.000000 3.872983 1.000000 3.316625- Create a vector 2, 4, 6, … , 18, 20 by two other ways.
The more ways to solve a problems you know the better.
(1:20)[c(FALSE,TRUE)]## [1] 2 4 6 8 10 12 14 16 18 20#(1:10)*220.6 Missing values
- Create a vector
vec4with values 300, 15, 8, 2, 0, 1, 110:
vec4 <- c(300, 15, 8, 20, 0, 1, 110)
vec4## [1] 300 15 8 20 0 1 110- Replace all values of the vector
vec4that are greater than 20 withNA.
vec4[vec4 > 20] <- NA- Check the vector
vec4:
vec4## [1] NA 15 8 20 0 1 NA- Sum over the vector
vec4using functionsum(). The answerNAis not a valid answer!
sum(vec4, na.rm = TRUE)## [1] 4420.7 Matrix
- Create a 4x4 matrix
M1containing ones.
M1 <- matrix(rep(1, 16), ncol = 4)
M1## [,1] [,2] [,3] [,4]
## [1,] 1 1 1 1
## [2,] 1 1 1 1
## [3,] 1 1 1 1
## [4,] 1 1 1 1- Replace all values of M1 that are not on the border (i.e. on positions [2,2], [2,3], [3,2] and [3,3]) with 2.
M1[2:3, 2:3] <- 2
M1## [,1] [,2] [,3] [,4]
## [1,] 1 1 1 1
## [2,] 1 2 2 1
## [3,] 1 2 2 1
## [4,] 1 1 1 1- Return the second and the third columns of the matrix
M1.
M1[,2:3]## [,1] [,2]
## [1,] 1 1
## [2,] 2 2
## [3,] 2 2
## [4,] 1 1- Compare (
==) the second column and the second row of the matrixM1.
M1[,2] == M1[2,]## [1] TRUE TRUE TRUE TRUE- *Create the multiplication table (9х9) as a matrix
mult_tab.
mult_tab <- matrix(rep(1:9, rep(9,9))*(1:9), nrow = 9)
mult_tab## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9]
## [1,] 1 2 3 4 5 6 7 8 9
## [2,] 2 4 6 8 10 12 14 16 18
## [3,] 3 6 9 12 15 18 21 24 27
## [4,] 4 8 12 16 20 24 28 32 36
## [5,] 5 10 15 20 25 30 35 40 45
## [6,] 6 12 18 24 30 36 42 48 54
## [7,] 7 14 21 28 35 42 49 56 63
## [8,] 8 16 24 32 40 48 56 64 72
## [9,] 9 18 27 36 45 54 63 72 81#Еще
#outer(1:9, 1:9, "*")
#1:9 %o% 1:9- *Select a smaller matrix from the matrix
mult_tabthat contains only rows from 6 to 8 and columns from 3 to 7.
mult_tab[6:8, 3:7]## [,1] [,2] [,3] [,4] [,5]
## [1,] 18 24 30 36 42
## [2,] 21 28 35 42 49
## [3,] 24 32 40 48 56- *Create a logical matrix with
TRUEif there is a two-digit number on a corresponding position of the matrixmult_tabandFALSEotherwise.
Matrix is basically a vector. You can work with a matrix the same way you work with a vector — with the only one index.
mult_tab >= 10## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9]
## [1,] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
## [2,] FALSE FALSE FALSE FALSE TRUE TRUE TRUE TRUE TRUE
## [3,] FALSE FALSE FALSE TRUE TRUE TRUE TRUE TRUE TRUE
## [4,] FALSE FALSE TRUE TRUE TRUE TRUE TRUE TRUE TRUE
## [5,] FALSE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE
## [6,] FALSE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE
## [7,] FALSE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE
## [8,] FALSE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE
## [9,] FALSE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE- *Create a matrix
mult_tab2from the matrixmult_tabwhere all values that are less than 0 replaced with 10.
mult_tab2 <- mult_tab
mult_tab2[mult_tab < 10] <- 0
mult_tab2## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9]
## [1,] 0 0 0 0 0 0 0 0 0
## [2,] 0 0 0 0 10 12 14 16 18
## [3,] 0 0 0 12 15 18 21 24 27
## [4,] 0 0 12 16 20 24 28 32 36
## [5,] 0 10 15 20 25 30 35 40 45
## [6,] 0 12 18 24 30 36 42 48 54
## [7,] 0 14 21 28 35 42 49 56 63
## [8,] 0 16 24 32 40 48 56 64 72
## [9,] 0 18 27 36 45 54 63 72 8120.8 List
Create a list list1:
list1 = list(numbers = 1:5, letters = letters, logic = TRUE)
list1## $numbers
## [1] 1 2 3 4 5
##
## $letters
## [1] "a" "b" "c" "d" "e" "f" "g" "h" "i" "j" "k" "l" "m" "n" "o" "p" "q" "r" "s"
## [20] "t" "u" "v" "w" "x" "y" "z"
##
## $logic
## [1] TRUE- Find the first element of the list
list1. The answer must be a list with length 1.
list1[1]## $numbers
## [1] 1 2 3 4 5- Extract the content of the first element of
list1by two different ways. The answer must be a vector.
list1[[1]]## [1] 1 2 3 4 5list1$numbers## [1] 1 2 3 4 5- Find the first value from the first item of the list
list1. The answer must be a vector.
list1[[1]][1]## [1] 1- Create a list
list2, that contains two listslist1. One of them must be namedpupa, the second one —lupa.
list2 = list(pupa = list1, lupa = list1)
list2## $pupa
## $pupa$numbers
## [1] 1 2 3 4 5
##
## $pupa$letters
## [1] "a" "b" "c" "d" "e" "f" "g" "h" "i" "j" "k" "l" "m" "n" "o" "p" "q" "r" "s"
## [20] "t" "u" "v" "w" "x" "y" "z"
##
## $pupa$logic
## [1] TRUE
##
##
## $lupa
## $lupa$numbers
## [1] 1 2 3 4 5
##
## $lupa$letters
## [1] "a" "b" "c" "d" "e" "f" "g" "h" "i" "j" "k" "l" "m" "n" "o" "p" "q" "r" "s"
## [20] "t" "u" "v" "w" "x" "y" "z"
##
## $lupa$logic
## [1] TRUE- *Extract the first item of the
list2, then extract the second item from this item, and then extract the third value of it.
list2[[1]][[2]][3]## [1] "c"20.9 Датафрейм
- Запустите команду
data(mtcars)чтобы загрузить встроенный датафрейм с информацией про автомобили. Каждая строчка датафрейма - модель автомобиля, каждая колонка - отдельная характеристика. Подробнее см.?mtcars.
data(mtcars)
mtcars- Изучите структуру датафрейма
mtcarsс помощью функцииstr().
str(mtcars)## 'data.frame': 32 obs. of 11 variables:
## $ mpg : num 21 21 22.8 21.4 18.7 18.1 14.3 24.4 22.8 19.2 ...
## $ cyl : num 6 6 4 6 8 6 8 4 4 6 ...
## $ disp: num 160 160 108 258 360 ...
## $ hp : num 110 110 93 110 175 105 245 62 95 123 ...
## $ drat: num 3.9 3.9 3.85 3.08 3.15 2.76 3.21 3.69 3.92 3.92 ...
## $ wt : num 2.62 2.88 2.32 3.21 3.44 ...
## $ qsec: num 16.5 17 18.6 19.4 17 ...
## $ vs : num 0 0 1 1 0 1 0 1 1 1 ...
## $ am : num 1 1 1 0 0 0 0 0 0 0 ...
## $ gear: num 4 4 4 3 3 3 3 4 4 4 ...
## $ carb: num 4 4 1 1 2 1 4 2 2 4 ...- Найдите значение третьей строчки четвертого столбца датафрейма
mtcars.
mtcars[3, 4]## [1] 93- Извлеките первые шесть строчек и первые шесть столбцов датафрейма
mtcars.
mtcars[1:6, 1:6]- Извлеките колонку
wtдатафреймаmtcars- массу автомобиля в тысячах фунтов.
mtcars$wt## [1] 2.620 2.875 2.320 3.215 3.440 3.460 3.570 3.190 3.150 3.440 3.440 4.070
## [13] 3.730 3.780 5.250 5.424 5.345 2.200 1.615 1.835 2.465 3.520 3.435 3.840
## [25] 3.845 1.935 2.140 1.513 3.170 2.770 3.570 2.780- Извлеките колонки из
mtcarsв следующем порядке:hp,mpg,cyl.
mtcars[, c("hp", "mpg", "cyl")]- Посчитайте количество автомобилей с 4 цилиндрами (
cyl) в датафреймеmtcars.
sum(mtcars$cyl == 4)## [1] 11- Посчитайте долю автомобилей с 4 цилиндрами (
cyl) в датафреймеmtcars.
mean(mtcars$cyl == 4)## [1] 0.34375- Найдите все автомобили мощностью не менее 100 лошадиных сил (
hp) в датафреймеmtcars.
mtcars[mtcars$hp >= 100, ]- Найдите все автомобили мощностью не менее 100 лошадиных сил (
hp) и 4 цилиндрами (cyl) в датафреймеmtcars.
mtcars[mtcars$hp >= 100 & mtcars$cyl == 4, ]- Посчитайте максимальную массу (
wt) автомобиля в выборке, воспользовавшись функциейmax():
max(mtcars$wt)## [1] 5.424- Посчитайте минимальную массу (
wt) автомобиля в выборке, воспользовавшись функциейmin():
min(mtcars$wt)## [1] 1.513- Найдите строчку датафрейма
mtcarsс самым легким автомобилем.
mtcars[mtcars$wt == min(mtcars$wt), ]- Извлеките строчки датафрейма
mtcarsс автомобилями, масса которых ниже средней массы.
mtcars[mtcars$wt < mean(mtcars$wt), ]- Масса автомобиля указана в тысячах фунтов. Создайте колонку
wt_kgс массой автомобиля в килограммах. Результат округлите до целых значений с помощью функцииround().
1 фунт = 0.45359237 кг.
mtcars$wt_kg <- round(mtcars$wt * 1000 * 0.45359237)
mtcars20.10 Условные конструкции
- Создайте вектор
vec5:
vec5 <- c(5, 20, 30, 0, 2, 9)- Создайте новый строковый вектор, где на месте чисел больше 10 в
vec5будет стоять “большое число,” а на месте остальных чисел — “маленькое число.”
ifelse(vec5 > 10, "большое число", "маленькое число")## [1] "маленькое число" "большое число" "большое число" "маленькое число"
## [5] "маленькое число" "маленькое число"- Загрузите файл heroes_information.csv в переменную
heroes.
heroes <- read.csv("data/heroes_information.csv",
stringsAsFactors = FALSE,
na.strings = c("-", "-99"))- Создайте новою колонку
hairвheroes, в которой будет значение"Bold"для тех супергероев, у которых в колонкеHair.colorстоит"No Hair", и значение"Hairy"во всех остальных случаях.
heroes$hair <- ifelse(heroes$Hair.color == "No Hair", "Bold", "Hairy")
head(heroes)- Создайте новою колонку
tallвheroes, в которой будет значение"tall"для тех супергероев, у которых в колонкеHeightстоит число больше 190, значение"short"для тех супергероев, у которых в колонкеHeightстоит число меньше 170, и значение"middle"во всех остальных случаях.
# heroes$tall <- dplyr::case_when(
# heroes$Height > 190 ~ "tall",
# heroes$Height < 170 ~ "short",
# TRUE ~ "middle"
# )
heroes$tall <- ifelse(heroes$Height > 190,
"tall",
ifelse(heroes$Height < 170,
"short",
"middle"))20.11 Создание функций
- Создайте функцию
plus_one(), которая принимает число и возвращает это же число + 1.
plus_one <- function(x) x + 1- Проверьте функцию
plus_one()на числе 41.
plus_one(41)## [1] 42- Создайте функцию
circle_area(), которая вычисляет площадь круга по радиусу согласно формуле \(\pi r^2\).
circle_area <- function(r) pi * r ^ 2- Посчитайте площадь круга с радиусом 5.
circle_area(5)## [1] 78.53982- Создайте функцию
cels2fahr(), которая будет превращать градусы по Цельсию в градусы по Фаренгейту.
cels2fahr <- function(x) x * 9 / 5 + 32- Проверьте на значениях -100, -40 и 0, что функция
cels2fahr()работает корректно.
cels2fahr(c(-100, -40, 0))## [1] -148 -40 32- Напишите функцию
highlight(), которая принимает на входе строковый вектор, а возвращает тот же вектор, но дополненный значением"***"в начале и конце вектора. Лучше всего это рассмотреть на примере:
highlight <- function(x) c("***", x, "***")highlight(c("Я", "Бэтмен!"))## [1] "***" "Я" "Бэтмен!" "***"- Теперь сделайте функцию
highlightболее гибкой. Добавьте в нее параметрwrapper =, который по умолчанию равен"***". Значение параметраwrapper =и будет вставлено в начало и конец вектора.
highlight <- function(x, wrapper = "***") c(wrapper, x, wrapper)- Проверьте написанную функцию на векторе
c("Я", "Бэтмен!").
highlight(c("Я", "Бэтмен!")) ## [1] "***" "Я" "Бэтмен!" "***"highlight(c("Я", "Бэтмен!"), wrapper = "__") ## [1] "__" "Я" "Бэтмен!" "__"- Создайте функцию
trim(), которая будет возвращать вектор без первого и последнего значения (вне зависимости от типа данных).
trim <- function(x) x[c(-1, -length(x))]- Проверьте, что функция
trim()работает корректно:
trim(1:7)## [1] 2 3 4 5 6trim(letters)## [1] "b" "c" "d" "e" "f" "g" "h" "i" "j" "k" "l" "m" "n" "o" "p" "q" "r" "s" "t"
## [20] "u" "v" "w" "x" "y"- Теперь добавьте в функцию
trim()параметрn =со значением по умолчанию 1. Этот параметр будет обозначать сколько значений нужно отрезать слева и справа от вектора.
trim <- function(x, n = 1) x[c(-1:-n, (-length(x)+n-1):-length(x))]- Проверьте полученную функцию:
trim(letters)## [1] "b" "c" "d" "e" "f" "g" "h" "i" "j" "k" "l" "m" "n" "o" "p" "q" "r" "s" "t"
## [20] "u" "v" "w" "x" "y"trim(letters, n = 2)## [1] "c" "d" "e" "f" "g" "h" "i" "j" "k" "l" "m" "n" "o" "p" "q" "r" "s" "t" "u"
## [20] "v" "w" "x"- Сделайте так, чтобы функция
trim()работала корректно сn = 0, т.е. функция возвращала бы исходный вектор без изменений.
trim <- function(x, n = 1) {
if (n == 0) return(x)
x[c(-1:-n, (-length(x)+n-1):-length(x))]
}trim(letters, n = 0)## [1] "a" "b" "c" "d" "e" "f" "g" "h" "i" "j" "k" "l" "m" "n" "o" "p" "q" "r" "s"
## [20] "t" "u" "v" "w" "x" "y" "z"- *Теперь добавьте проверку на адекватность входных данных: функция
trim()должна выдавать ошибку, еслиn =меньше нуля или еслиn =слишком большой и отрезает все значения вектора:
trim <- function(x, n = 1) {
if (n < 0) stop("n не может быть меньше нуля!")
l <- length(x)
if (n > ceiling(l/2) - 1) stop("n слишком большой!")
if (n == 0) return(x)
x[c(-1:-n, (-l+n-1):-l)]
}- *Проверьте полученную функцию
trim():
trim(1:6, 3)## Error in trim(1:6, 3): n слишком большой!trim(1:6, -1)## Error in trim(1:6, -1): n не может быть меньше нуля!- Создайте функцию
na_n(), которая будет возвращать количествоNAв векторе.
na_n <- function(x) sum(is.na(x))- Проверьте функцию
na_n()на векторе:
na_n(c(NA, 3:5, NA, 2, NA))## [1] 3- Напишите функцию
factors(), которая будет возвращать все делители числа в виде числового вектора.
Здесь может понадобиться оператор для получения остатка от деления:
%%.
factors <- function(x) (1:x)[x %% (1:x) == 0]- Проверьте функцию
factors()на простых и сложных числах:
factors(3)## [1] 1 3factors(161)## [1] 1 7 23 161factors(1984)## [1] 1 2 4 8 16 31 32 62 64 124 248 496 992 1984- *Напишите функцию
is_prime(), которая проверяет, является ли число простым.
Здесь может пригодиться функция
any()- она возвращаетTRUE, если в векторе есть хотя бы одинTRUE.
is_prime <- function(x) !any(x%%(2:(x-1)) == 0)
#is_prime <- function(x) length(factors(x)) == 2 #Используя уже написанную функцию factors()- Проверьте какие года были для нас простыми, а какие нет:
is_prime(2017)## [1] TRUEis_prime(2019)## [1] FALSE2019/3 #2019 делится на 3 без остатка## [1] 673is_prime(2020)## [1] FALSEis_prime(2021)## [1] FALSE- *Создайте функцию
monotonic(), которая возвращаетTRUE, если значения в векторе не убывают (то есть каждое следующее - больше или равно предыдущему) или не возврастают.
Полезная функция для этого —
diff()— возвращает разницу соседних значений.
monotonic <- function(x) all(diff(x)>=0) | all(diff(x)<=0)monotonic(1:7)## [1] TRUEmonotonic(c(1:5,5:1))## [1] FALSEmonotonic(6:-1)## [1] TRUEmonotonic(c(1:5, rep(5, 10), 5:10))## [1] TRUEБинарные операторы типа + или %in% тоже представляют собой функции. Более того, мы можем создавать свои бинарные операторы! В этом нет особой сложности — нужно все так же создавать функцию (для двух переменных), главное окружать их % и название обрамлять обратными штрихами `. Например, можно сделать свой бинарный оператор %notin%, который будет выдавать TRUE, если значения слева нет в векторе справа:
`%notin%` <- function(x, y) ! (x %in% y)
1:10 %notin% c(1, 4, 5)## [1] FALSE TRUE TRUE FALSE FALSE TRUE TRUE TRUE TRUE TRUE- *Создайте бинарный оператор
%without%, который будет возвращать все значения вектора слева без значений вектора справа.
`%without%` <- function(x, y) x[!x %in% y]c("а", "и", "б", "сидели", "на", "трубе") %without% c("а", "б")## [1] "и" "сидели" "на" "трубе"- *Создайте бинарный оператор
%between%, который будет возвращатьTRUE, если значение в векторе слева накходится в диапазоне значений вектора справа:
`%between%` <- function(x, y) x >= min(y) & x <= max(y)1:10 %between% c(1, 4, 5)## [1] TRUE TRUE TRUE TRUE TRUE FALSE FALSE FALSE FALSE FALSE20.12 Семейство функций apply()
- Создайте матрицу
M2:
M2 <- matrix(c(20:11, 11:20), nrow = 5)
M2## [,1] [,2] [,3] [,4]
## [1,] 20 15 11 16
## [2,] 19 14 12 17
## [3,] 18 13 13 18
## [4,] 17 12 14 19
## [5,] 16 11 15 20- Посчитайте максимальное значение матрицы
M2по каждой строчке.
apply(M2, 1, max)## [1] 20 19 18 19 20- Посчитайте максимальное значение матрицы
M2по каждому столбцу.
apply(M2, 2, max)## [1] 20 15 15 20- Посчитайте среднее значение матрицы
M2по каждой строке.
apply(M2, 1, mean)## [1] 15.5 15.5 15.5 15.5 15.5- Посчитайте среднее значение матрицы
M2по каждому столбцу.
apply(M2, 2, mean)## [1] 18 13 13 18- Создайте список
list3:
list3 <- list(
a = 1:5,
b = 0:20,
c = 4:24,
d = 6:3,
e = 6:25
)- Найдите максимальное значение каждого вектора списка
list3.
sapply(list3, max)## a b c d e
## 5 20 24 6 25- Посчитайте сумму каждого вектора списка
list3.
sapply(list3, sum)## a b c d e
## 15 210 294 18 310- Посчитайте длину каждого вектора списка
list3.
sapply(list3, length)## a b c d e
## 5 21 21 4 20- Напишите функцию
max_item(), которая будет принимать на входе список, а возвращать - (первый) самый длинный его элемент.
Для этого вам может понадобиться функция
which.max(), которая возвращает индекс максимального значения (первого, если их несколько).
max_item <- function (x) x[[which.max(sapply(x, length))]]- Проверьте функцию
max_item()на спискеlist3.
max_item(list3)## [1] 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20- Теперь мы сделаем сложный список
list4:
list4 <- list(1:3, 3:40, list3)- Посчитайте длину каждого вектора в списке, в т.ч. для списка внутри. Результат должен быть списком с такой же структорой, как и изначальный список
list4.
Для этого может понадобиться функция
rapply(): recursive lapply
rapply(list4, length, how = "list")## [[1]]
## [1] 3
##
## [[2]]
## [1] 38
##
## [[3]]
## [[3]]$a
## [1] 5
##
## [[3]]$b
## [1] 21
##
## [[3]]$c
## [1] 21
##
## [[3]]$d
## [1] 4
##
## [[3]]$e
## [1] 20- *Загрузите набор данных
heroesи посчитайте, сколькоNAв каждом из столбцов.
Для этого удобно использовать ранее написанную функцию
na_n().
sapply(heroes, na_n)## X name Gender Eye.color Race Hair.color Height
## 0 0 29 172 304 172 217
## Publisher Skin.color Alignment Weight hair tall
## 0 662 7 239 172 217- *Используя ранее написанную функцию
is_prime(), напишите функциюprime_numbers(), которая будет возвращать все простые числа до выбранного числа.
is_prime <- function(x) !any(x %% (2:(x - 1)) == 0)
prime_numbers <- function(x) (2:x)[sapply(2:x, is_prime)]prime_numbers(200)## [1] 3 5 7 11 13 17 19 23 29 31 37 41 43 47 53 59 61 67 71
## [20] 73 79 83 89 97 101 103 107 109 113 127 131 137 139 149 151 157 163 167
## [39] 173 179 181 191 193 197 19920.13 magrittr::%>%
- Rewrite the following sentences using pipe
%>%:
sqrt(sum(1:10))## [1] 7.4161981:10 %>%
sum() %>%
sqrt()abs(min(-5:5))## [1] 5-5:5 %>%
min() %>%
abs()c("Корень из", 2, "равен", sqrt(2))## [1] "Корень из" "2" "равен" "1.4142135623731"2 %>% c("Корень из", ., "равен", sqrt(.))20.14 Columns selection: dplyr::select()
For solving the next tasks you will need datasets heroes and powers that you can import using the following commands:
library(tidyverse)
heroes <- read_csv("https://raw.githubusercontent.com/Pozdniakov/tidy_stats/master/data/heroes_information.csv",
na = c("-", "-99"))
powers <- read_csv("https://raw.githubusercontent.com/Pozdniakov/tidy_stats/master/data/super_hero_powers.csv")- Select the first 4 columns from
powers.
powers %>%
select(1:4)- Select all columns from
ReflexestoEmpathyin the tibblepowers:
powers %>%
select(Reflexes:Empathy)- Select all columns from the tibble
powersexcept the first one (hero_names):
powers %>%
select(!hero_names)20.15 Rows selection: dplyr::slice() and dplyr::filter()
- Extract rows that contain information about superheroes which have weight more than 500 kg.
heroes %>%
filter(Weight > 500)- Extract rows that contain information about female superheroes which have weight more than 500 kg.
heroes %>%
filter(Weight > 500 & Gender == "Female")- Extract rows that contain information about human (race is equal to
"Human") female superheroes which have weight more than 500 kg. Select the first 5 rows from that.
heroes %>%
filter(Race == "Human" & Gender == "Female") %>%
slice(1:5)20.16 Rows sorting: dplyr::arrange()
- Select columns
name,Gender,Heightfor the tibbleheroesand sort them byHeightin increasing order.
heroes %>%
select(name, Gender, Height) %>%
arrange(Height)- Select columns
name,Gender,Heightfor the tibbleheroesand sort them byHeightin decreasing order.
heroes %>%
select(name, Gender, Height) %>%
arrange(desc(Height))- Select columns
name,Gender,Heightfor the tibbleheroesand sort them first byGender, then byHeightin decreasing order.
heroes %>%
select(name, Gender, Height) %>%
arrange(Gender, desc(Height))20.17 Unique values: dplyr::distinct()
- Exctract unique values from column
Eye colorinheroes.
heroes %>%
distinct(`Eye color`)- Exctract unique values from column
Hair colorinheroes.
heroes %>%
distinct(`Hair color`)20.18 Making new columns: dplyr::mutate() and dplyr::transmute()
- Create new column
height_mwith height of superheroes in meters, then selectnameandheight_mcolumns.
heroes %>%
mutate(height_m = Height/100) %>%
select(name, height_m)20.19 Агрегация: dplyr::group_by() %>% summarise()
- Calculate number of superheroes by races and sort it in decreasing order. Extract the first 5 rows.
heroes %>%
count(Race, sort = TRUE) %>%
slice(1:5)- Calculate the average height by gender.
heroes %>%
group_by(Gender) %>%
summarise(height_mean = mean(Height, na.rm = TRUE))20.20 Operations with several columns: across()
- Count number of
NAin each column, grouping byGender.
na_n <- function(x) sum(is.na(x))
heroes %>%
group_by(Gender) %>%
summarise(across(everything(), na_n))- Count number of
NAin each column that ends with"color", grouping byGender.
na_n <- function(x) sum(is.na(x))
heroes %>%
group_by(Gender) %>%
summarise(across(ends_with("color"), na_n))20.21 Merging dataframes: *_join {#solution_join}
- Create a tibble
web creatorssuch that it contains only superheroes that can create web, i.e. they haveTRUEin columnWeb Creationin tibblepowers.
powers_web <- powers %>%
select(hero_names, `Web Creation`)
web_creators <- left_join(heroes, powers_web, by = c("name" = "hero_names")) %>%
filter(`Web Creation`)
web_creators- Find all superheroes that can be found in
heroesbut are absent inpowers. The answer must be a character vector with superheroes names.
anti_join(heroes, powers, by = c("name" = "hero_names")) %>%
pull(name)## [1] "Agent 13" "Alfred Pennyworth" "Arsenal"
## [4] "Batgirl III" "Batgirl V" "Beetle"
## [7] "Black Goliath" "Black Widow II" "Blaquesmith"
## [10] "Bolt" "Boomer" "Box"
## [13] "Box III" "Captain Mar-vell" "Cat II"
## [16] "Cecilia Reyes" "Clea" "Clock King"
## [19] "Colin Wagner" "Colossal Boy" "Corsair"
## [22] "Cypher" "Danny Cooper" "Darkside"
## [25] "ERG-1" "Fixer" "Franklin Storm"
## [28] "Giant-Man" "Giant-Man II" "Goliath"
## [31] "Goliath" "Goliath" "Guardian"
## [34] "Hawkwoman" "Hawkwoman II" "Hawkwoman III"
## [37] "Howard the Duck" "Jack Bauer" "Jesse Quick"
## [40] "Jessica Sanders" "Jigsaw" "Jyn Erso"
## [43] "Kid Flash II" "Kingpin" "Meteorite"
## [46] "Mister Zsasz" "Mogo" "Moloch"
## [49] "Morph" "Nite Owl II" "Omega Red"
## [52] "Paul Blart" "Penance" "Penance I"
## [55] "Plastic Lad" "Power Man" "Renata Soliz"
## [58] "Ronin" "Shrinking Violet" "Snake-Eyes"
## [61] "Spider-Carnage" "Spider-Woman II" "Stacy X"
## [64] "Thunderbird II" "Two-Face" "Vagabond"
## [67] "Vision II" "Vulcan" "Warbird"
## [70] "White Queen" "Wiz Kid" "Wondra"
## [73] "Wyatt Wingfoot" "Yellow Claw"- Find all superheroes that can be found in
powersbut are absent inheroes. The answer must be a character vector with superheroes names.
anti_join(powers, heroes, by = c("hero_names" = "name")) %>%
pull(hero_names)## [1] "3-D Man" "Bananaman" "Bizarro-Girl"
## [4] "Black Vulcan" "Blue Streak" "Bradley"
## [7] "Clayface" "Concrete" "Dementor"
## [10] "Doctor Poison" "Fire" "Hellgramite"
## [13] "Lara Croft" "Little Epic" "Lord Voldemort"
## [16] "Orion" "Peek-a-Boo" "Queen Hippolyta"
## [19] "Reactron" "SHDB" "Stretch Armstrong"
## [22] "TEST" "Tommy Clarke" "Tyrant"20.22 Tidy data
First, create a tibble heroes_weight by copying the following code:
heroes_weight <- heroes %>%
filter(Publisher %in% c("DC Comics", "Marvel Comics")) %>%
group_by(Gender, Publisher) %>%
summarise(weight_mean = mean(Weight, na.rm = TRUE)) %>%
drop_na()
heroes_weight - Turn the tibble
heroes_weightinto wide tibble.
heroes_weight %>%
pivot_wider(names_from = "Publisher", values_from = "weight_mean")- Then, convert it back to the long format:
heroes_weight %>%
pivot_wider(names_from = "Publisher", values_from = "weight_mean") %>%
pivot_longer(cols = !Gender,
names_to = "Publisher",
values_to = "weight_mean")- Turn the tibble
powersinto a long tibble with three columns:hero_names,power(superpower names) andhas(does this superhero have this ability?).
powers %>%
pivot_longer(cols = !hero_names,
names_to = "power",
values_to = "has")- Turn the tibble
powersinto wide form again but with the new structure: each row is a superpower, each column is a superhero (except for the first column - superpower name).
powers %>%
pivot_longer(cols = !hero_names,
names_to = "power",
values_to = "has") %>%
pivot_wider(names_from = hero_names,
values_from = has)20.23 Описательная статистика
Для выполнения задания создайте вектор height из колонки Height датасета heroes, удалив в нем NA.
height <- heroes %>%
drop_na(Height) %>%
pull(Height)- Посчитайте среднее в векторе
height.
mean(height)## [1] 186.7263- Посчитайте усеченное среднее в векторе
heightс усечением 5% значений с обоих сторон.
mean(height, trim = 0.05)## [1] 182.5846- Посчитайте медиану в векторе
height.
median(height)## [1] 183- Посчитайте стандартное отклонение в векторе
height.
sd(height)## [1] 59.25189- Посчитайте межквартильный размах в векторе
height.
IQR(height)## [1] 18- Посчитайте ассиметрию в векторе
height.
psych::skew(height)## [1] 8.843432Посчитайте эксцесс в векторе height.
psych::kurtosi(height)## [1] 105.0297Примените функции для получения множественных статистик на векторе height.
summary(height)## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 15.2 173.0 183.0 186.7 191.0 975.0psych::describe(height)skimr::skim(height)| Name | height |
| Number of rows | 517 |
| Number of columns | 1 |
| _______________________ | |
| Column type frequency: | |
| numeric | 1 |
| ________________________ | |
| Group variables | None |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 | hist |
|---|---|---|---|---|---|---|---|---|---|---|
| data | 0 | 1 | 186.73 | 59.25 | 15.2 | 173 | 183 | 191 | 975 | ▇▁▁▁▁ |
20.24 Построение графиков в ggplot2
- Нарисуйте столбиковую диаграмму (
geom_bar()), которая будет отражать количество супергероев издателей"Marvel Comics","DC Comics"и всех остальных (отдельным столбиком) из датасетаheroes.
heroes %>%
mutate(Publisher = ifelse(Publisher %in% c("Marvel Comics", "DC Comics"),
Publisher,
"Other publishers")) %>%
#mutate(Publisher = fct_lump(Publisher, 2)) %>% #Еще один способ сделать то же самое, но через forcats
ggplot(aes(x = Publisher)) +
geom_bar()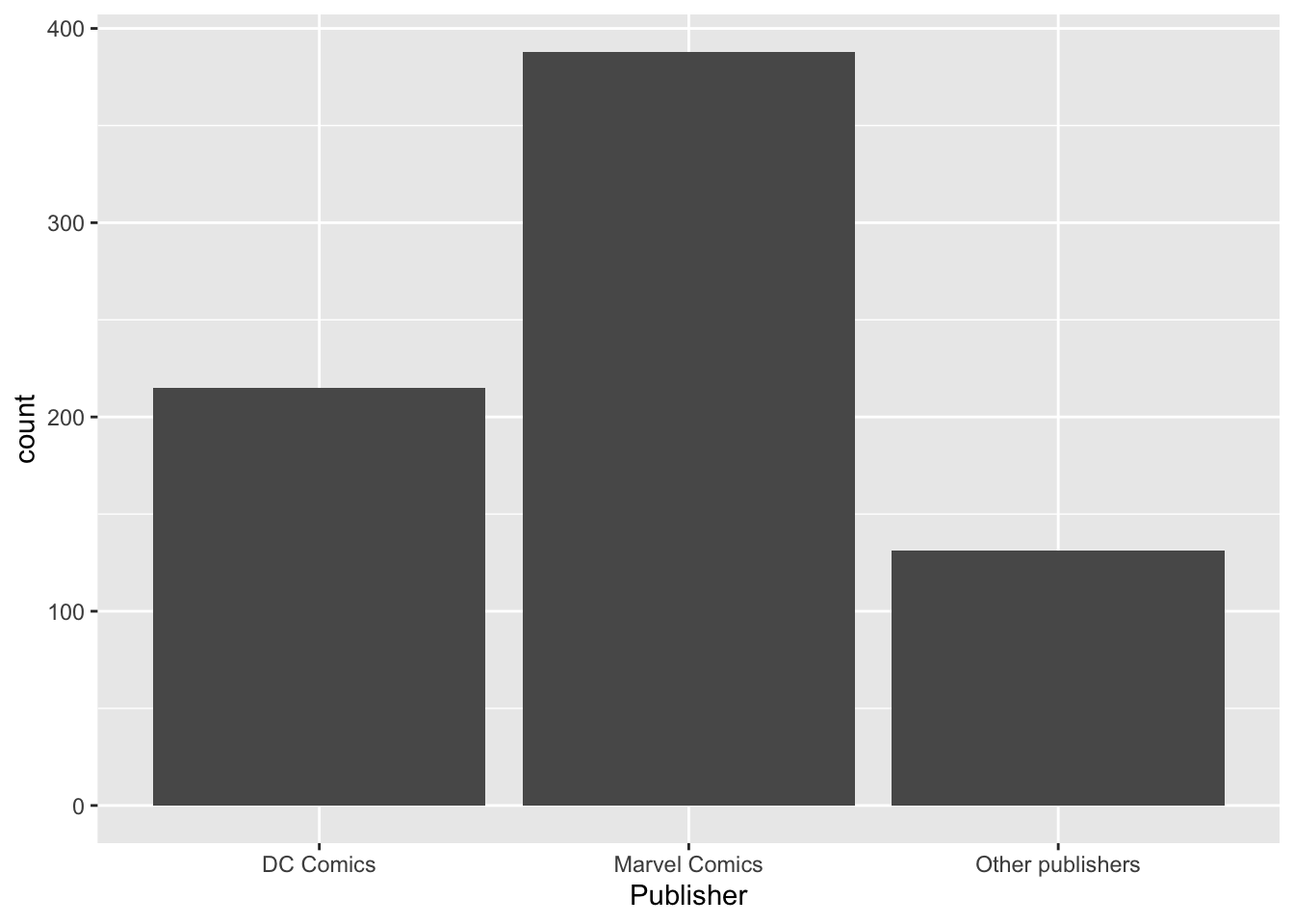
- Добавьте к этой диаграме заливку цветом (
fill =) в зависимости от распределенияGenderвнутри каждой группы.
heroes %>%
mutate(Publisher = ifelse(Publisher %in% c("Marvel Comics", "DC Comics"),
Publisher,
"Other publishers")) %>%
#mutate(Publisher = fct_lump(Publisher, 2)) %>% #Еще один способ сделать то же самое, но через forcats
ggplot(aes(x = Publisher, fill = Gender)) +
geom_bar()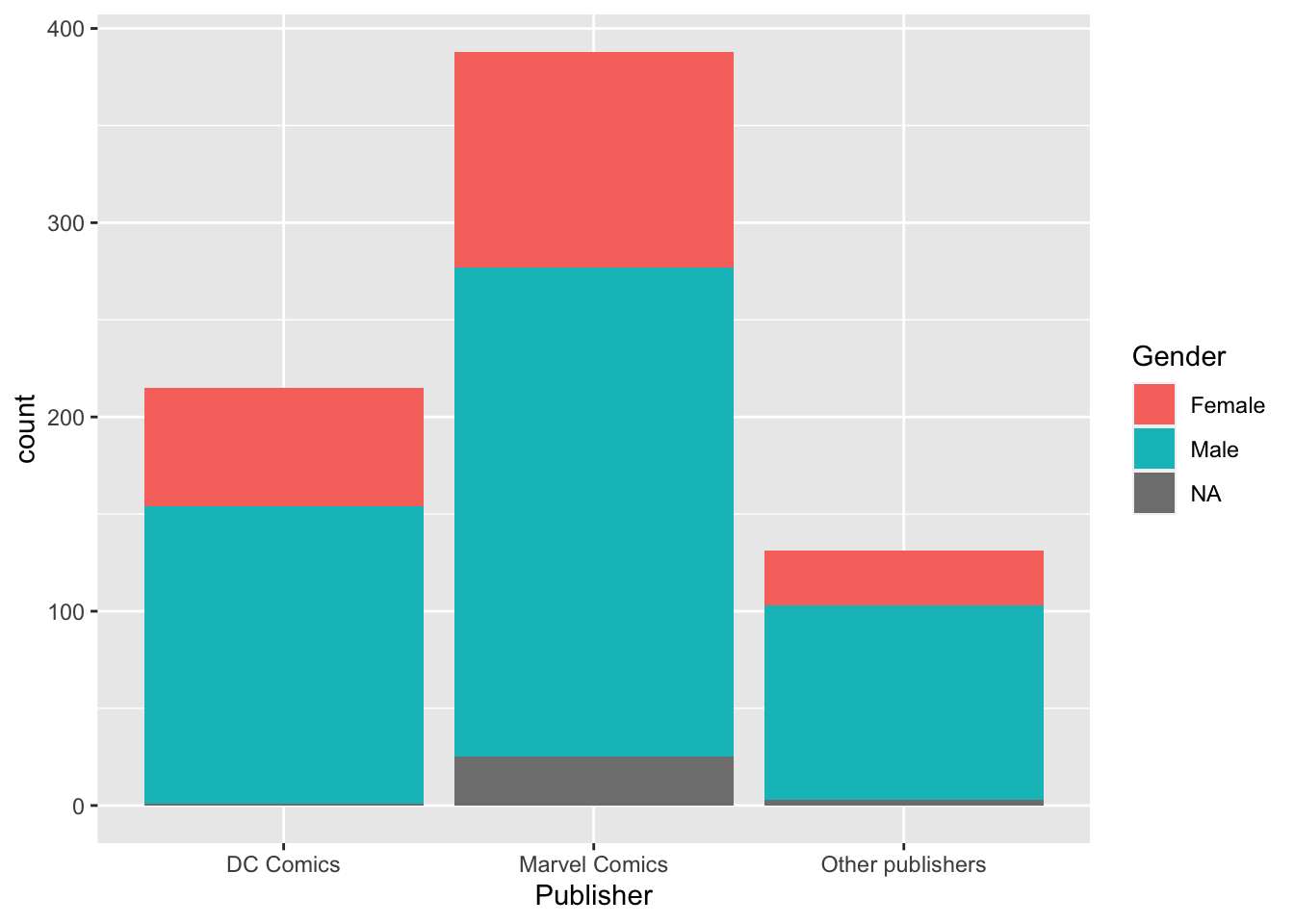
- Сделайте так, чтобы каждый столбик был максимальной высоты (
position = "fill").
heroes %>%
mutate(Publisher = ifelse(Publisher %in% c("Marvel Comics", "DC Comics"),
Publisher,
"Other publishers")) %>%
#mutate(Publisher = fct_lump(Publisher, 2)) %>% #Еще один способ сделать то же самое, но через forcats
ggplot(aes(x = Publisher, fill = Gender)) +
geom_bar(position = "fill")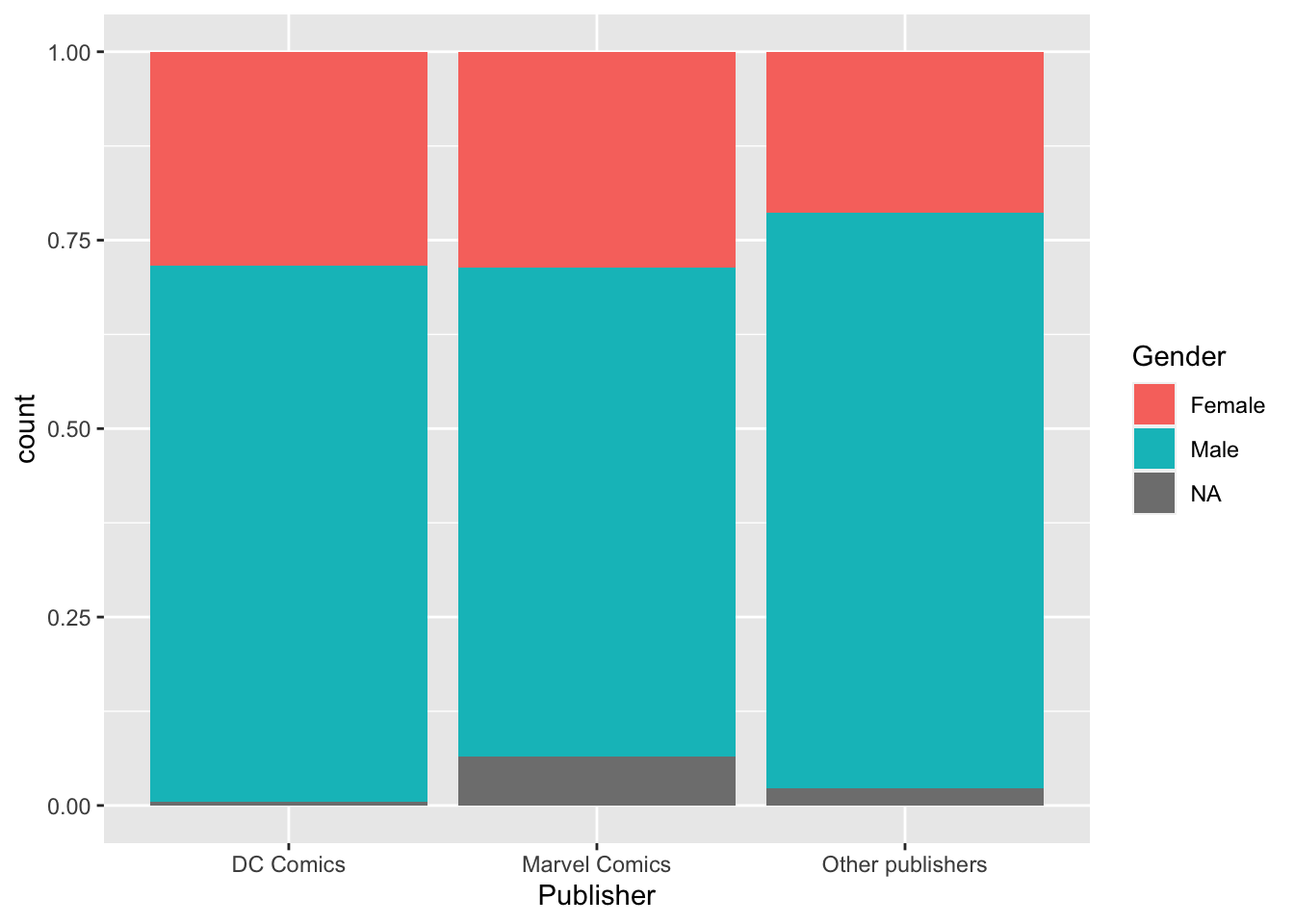
- Финализируйте график, задав ему описания осей (например, функция
labs()), использовав процентную шкалу (scale_y_continuous(labels = scales::percent)) и задав темуtheme_minimal().
heroes %>%
mutate(Publisher = ifelse(Publisher %in% c("Marvel Comics", "DC Comics"),
Publisher,
"Other publishers")) %>%
#mutate(Publisher = fct_lump(Publisher, 2)) %>% #Еще один способ сделать то же самое, но через forcats
ggplot(aes(x = Publisher, fill = Gender)) +
geom_bar(position = "fill") +
labs(title = "Распределение супергероев по полу у разных издателей коммиксов",
x = "Издатель",
y = "Количество супергероев")+
scale_y_continuous(labels = scales::percent) +
theme_minimal()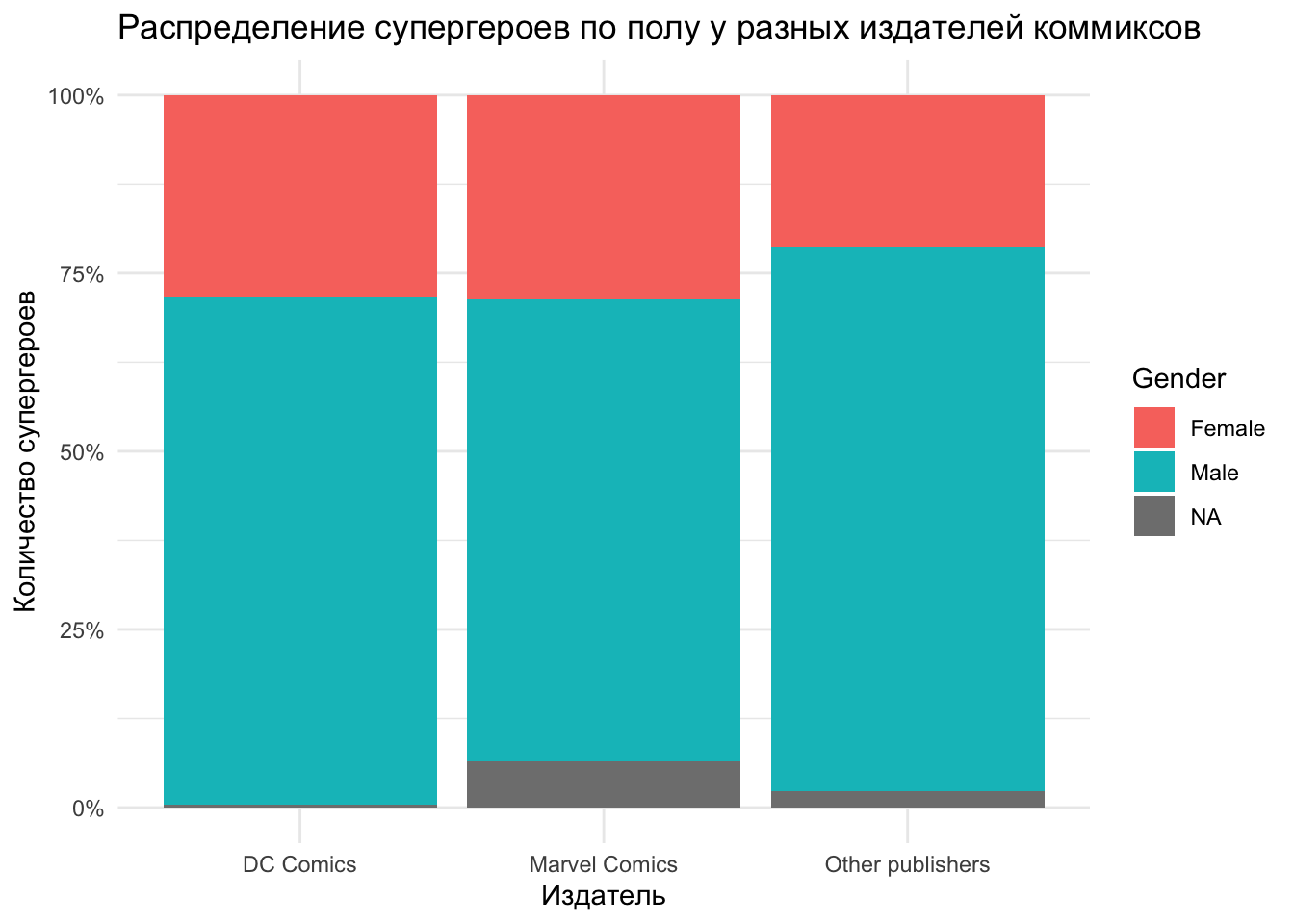
Создайте диаграмму рассеяния для датасета heroes, для которой координаты по оси x будут взяты из колонки Height, а координаты по оси y — из колонки Weight.
heroes %>%
ggplot(aes(x = Height, y = Weight)) +
geom_point()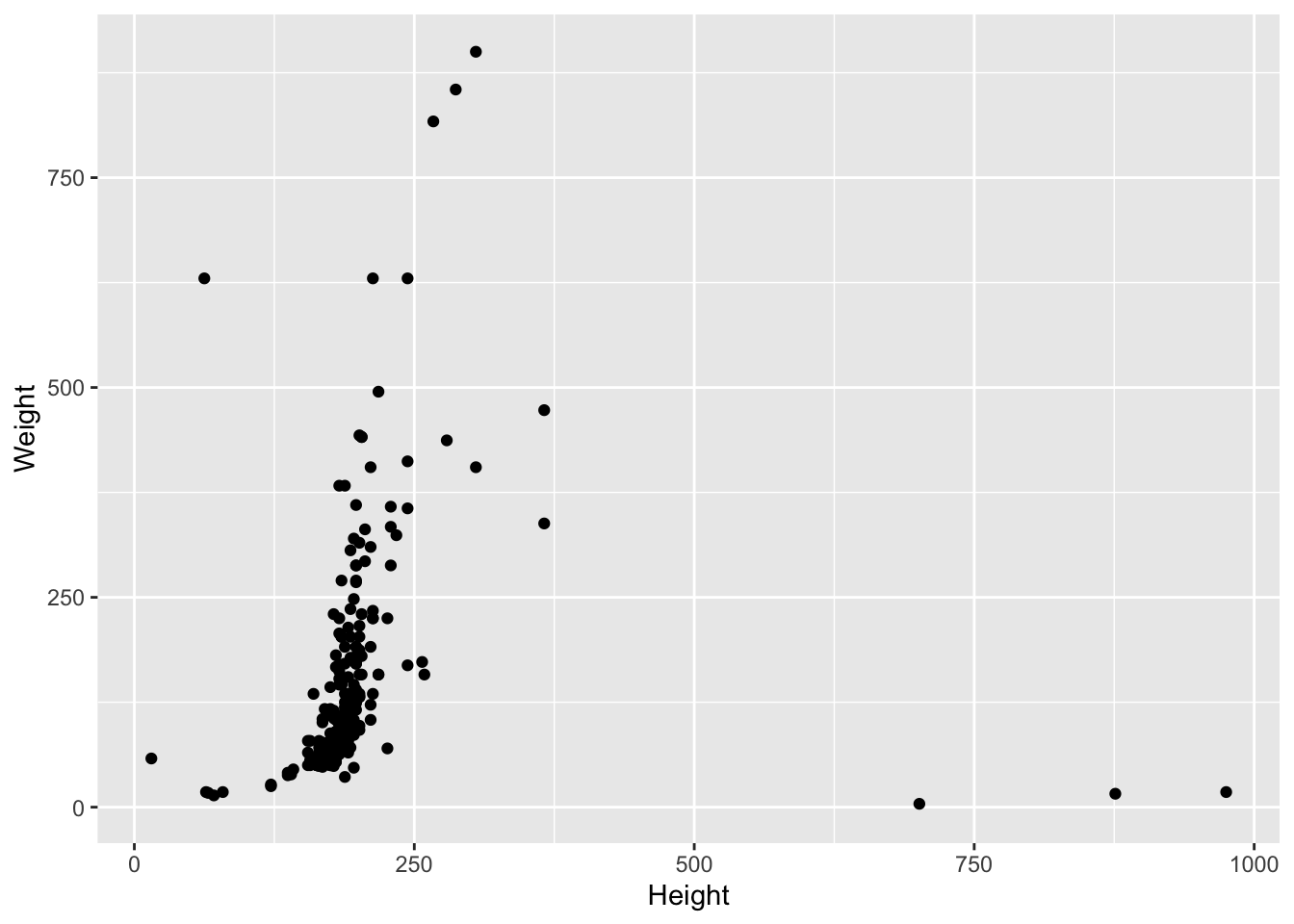
- Удалите с графика все экстремальные значения, для которых
Weightбольше или равен 700 илиHeightбольше или равен 400. (Подсказка: это можно делать как средствамиggplot2, так и функциейfilter()изdplyr).
heroes %>%
filter(Weight < 700 & Height < 400) %>%
ggplot(aes(x = Height, y = Weight)) +
geom_point()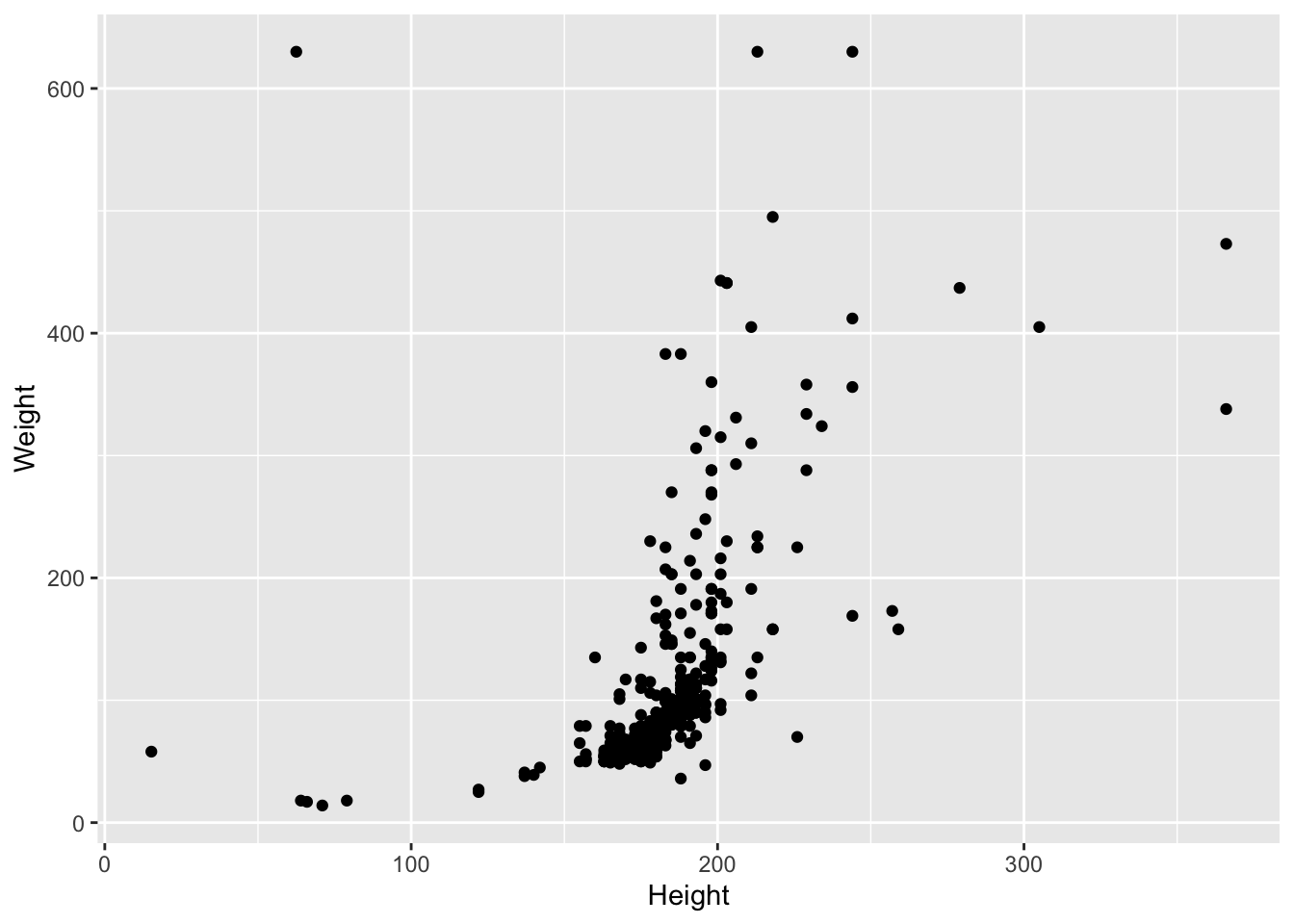
- Раскрасьте точки в зависимости от
Gender, сделайте их полупрозрачными ( параметрalpha =).
heroes %>%
filter(Weight < 700 & Height < 400) %>%
ggplot(aes(x = Height, y = Weight)) +
geom_point(aes(colour = Gender), alpha = 0.5)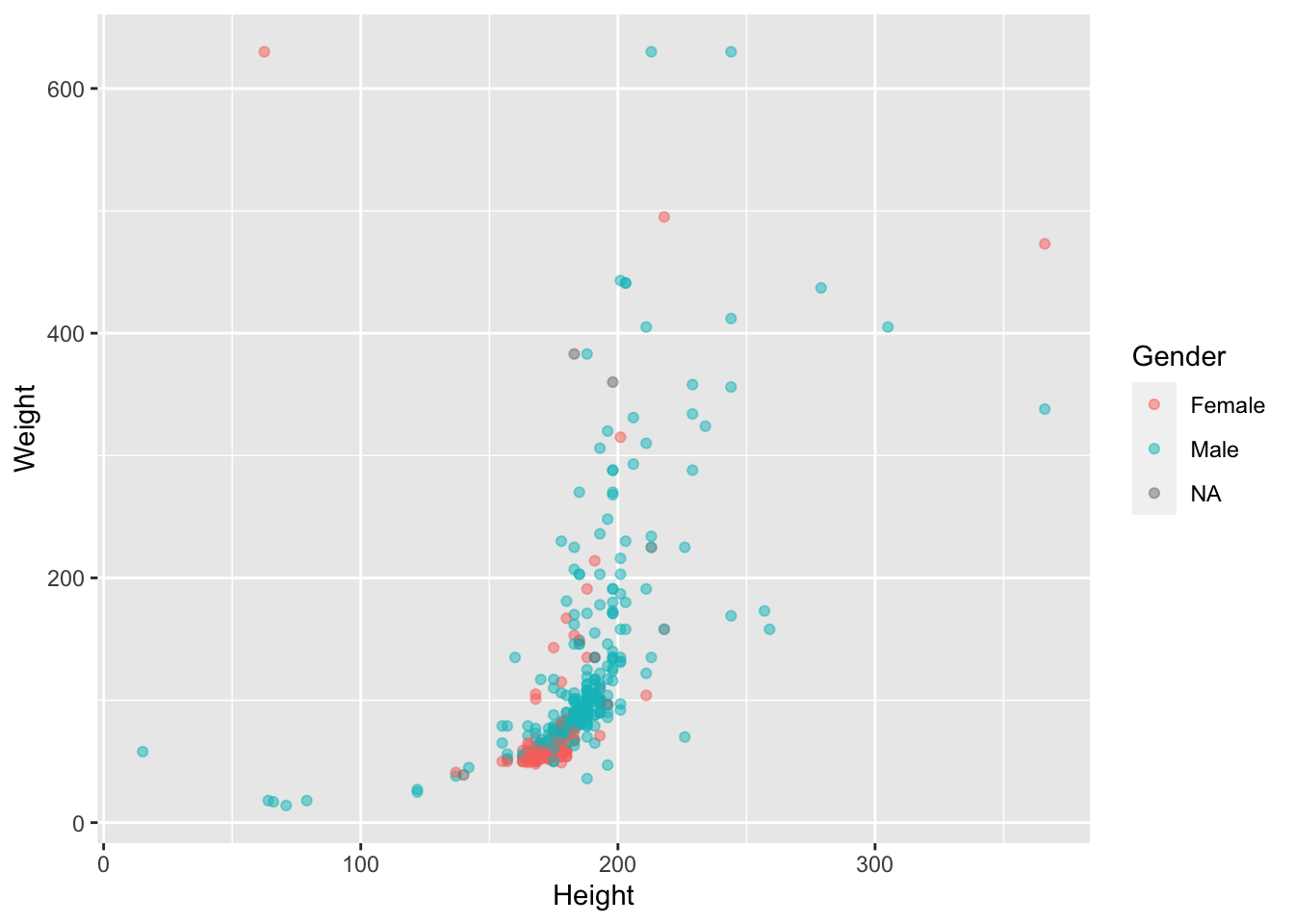
- Сделайте так, чтобы координатная плоскость имела соотношение 1:1 шкал по оси x и y. Этого можно добиться с помощью функции
coord_fixed().
heroes %>%
filter(Weight < 700 & Height < 400) %>%
ggplot(aes(x = Height, y = Weight)) +
geom_point(aes(colour = Gender), alpha = 0.5) +
coord_fixed()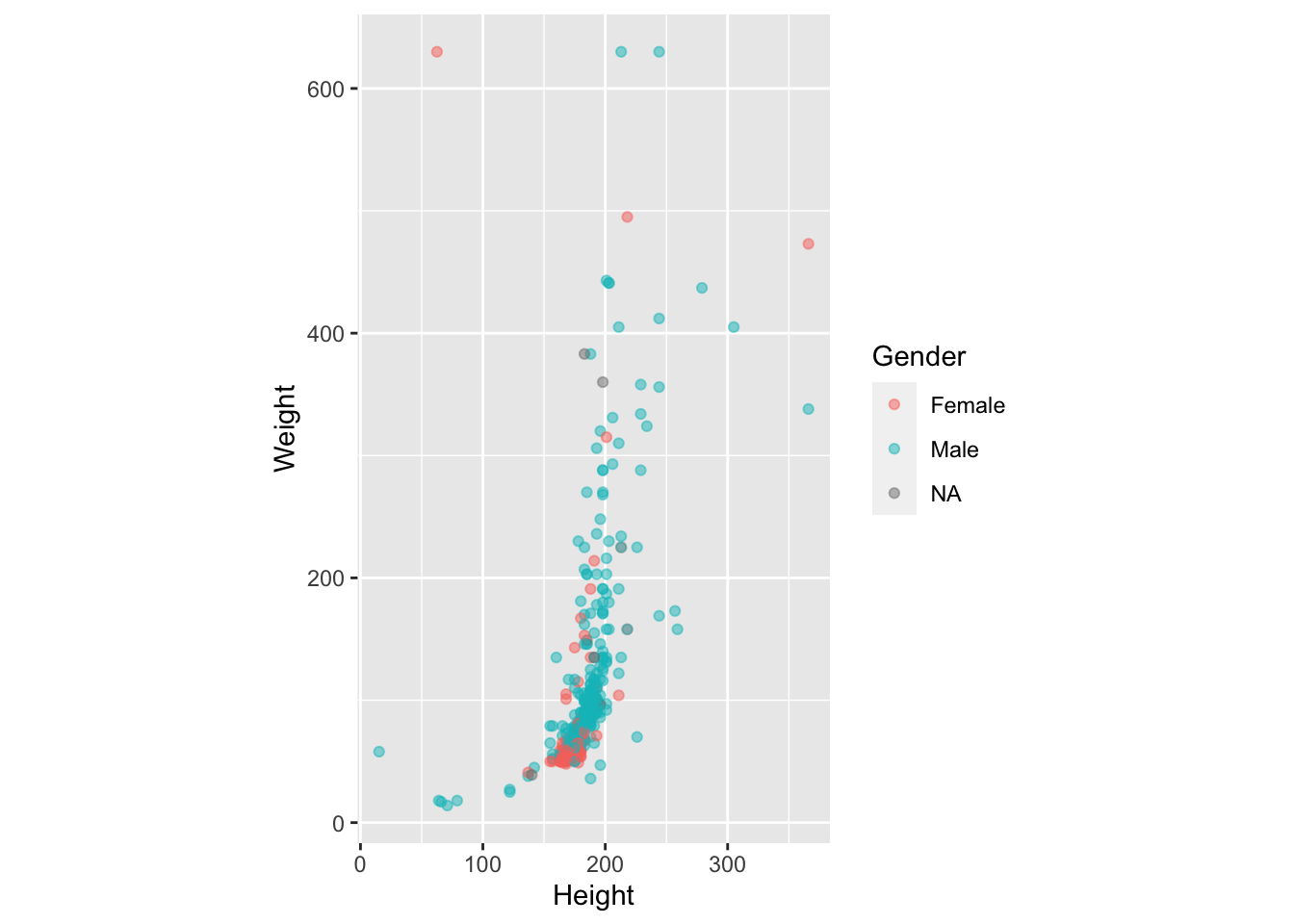
Разделите график (facet_wrap()) на три: для "DC Comics","Marvel Comics" и всех остальных.
heroes %>%
mutate(Publisher = ifelse(Publisher %in% c("Marvel Comics", "DC Comics"),
Publisher,
"Other publishers")) %>%
filter(Weight < 700 & Height < 400) %>%
ggplot(aes(x = Height, y = Weight)) +
geom_point(aes(colour = Gender), alpha = 0.5) +
coord_fixed() +
facet_wrap(~Publisher)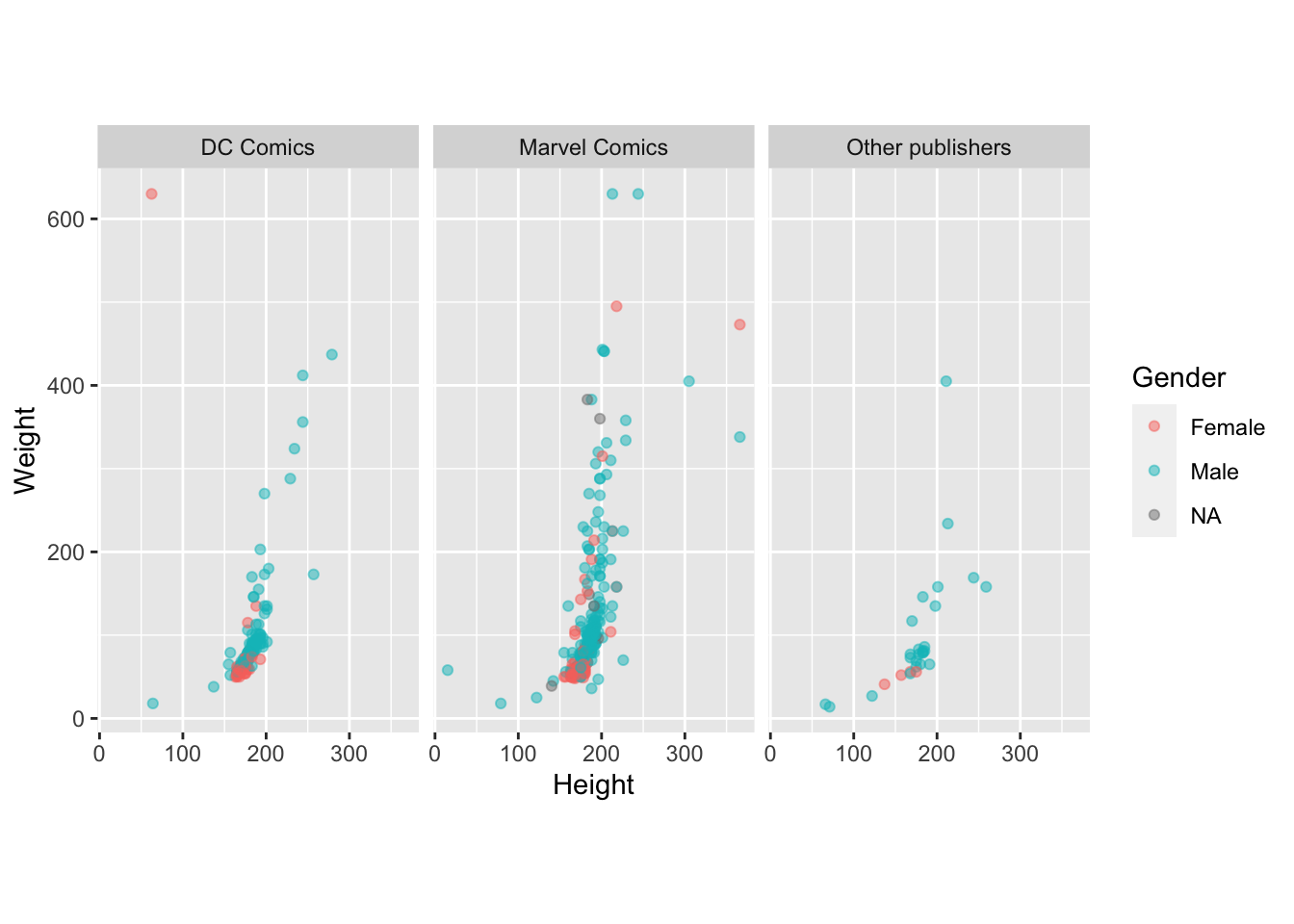
- Используйте для графика тему
theme_linedraw().
heroes %>%
mutate(Publisher = ifelse(Publisher %in% c("Marvel Comics", "DC Comics"),
Publisher,
"Other publishers")) %>%
filter(Weight < 700 & Height < 400) %>%
ggplot(aes(x = Height, y = Weight)) +
geom_point(aes(colour = Gender), alpha = 0.5) +
coord_fixed() +
facet_wrap(~Publisher)+
theme_linedraw()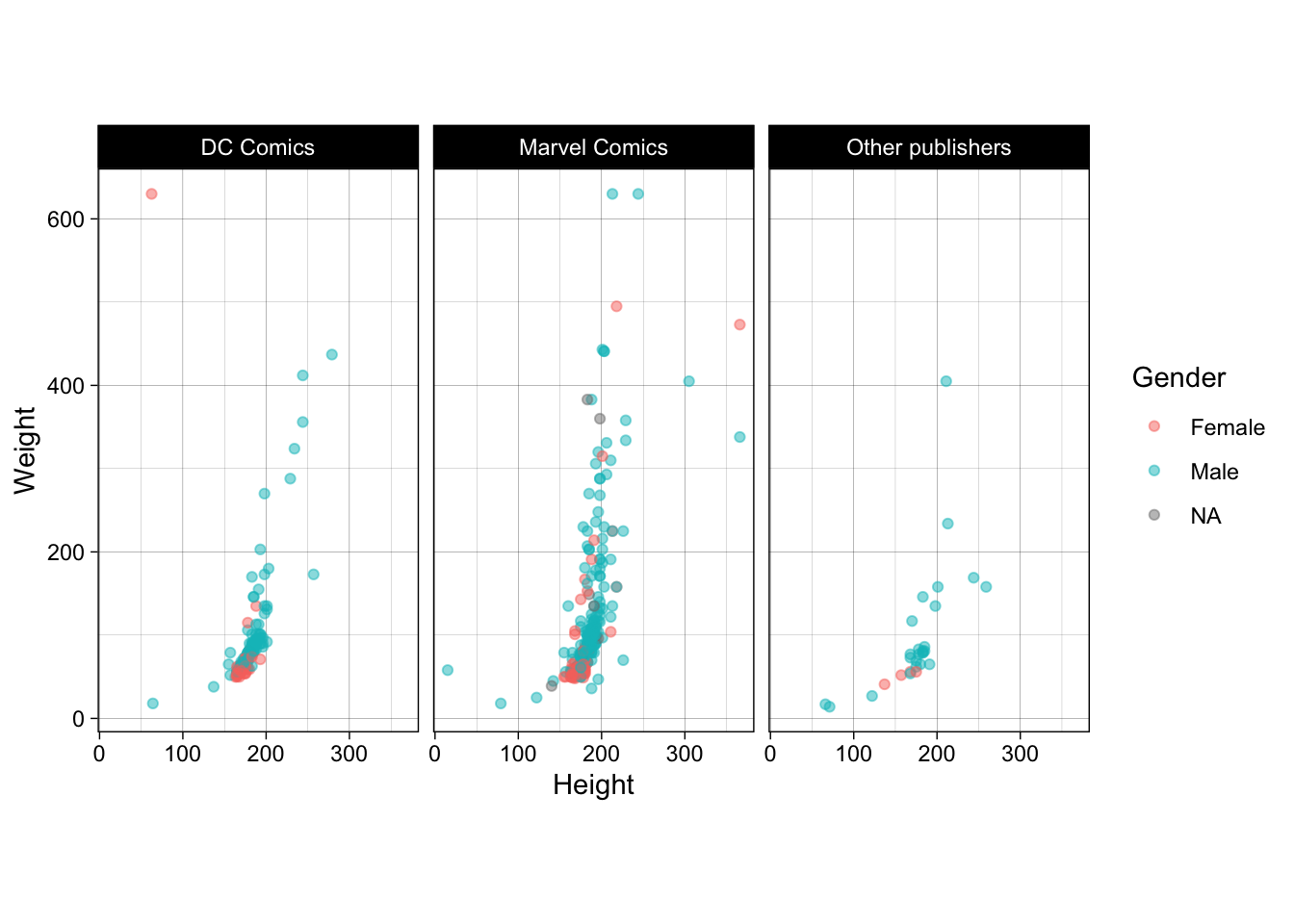
- Постройте новый график (или возьмите старый) по датасетам
heroesи/илиpowersи сделайте его некрасивым! Чем хуже у вас получится график, тем лучше. Желательно, чтобы этот график был по-прежнему графиком, а не произведением абстрактного искусства. Разница очень тонкая, но она есть.
- Постройте новый график (или возьмите старый) по датасетам
Вот несколько подсказок для этого задания:
Для вдохновения посмотрите на вот эти графики.
Для реально плохих графиков вам придется покопаться с настройками темы. Посмотрите подсказку по темам
?theme, попытайтесь что-то поменять в теме.Экспериментируйте с разными геомами и необычными их применениями.
По изучайте дополнения к
gpplot2.Попробуйте подготовить интересные данные для этого графика.
НЕТ ПРАВИЛЬНОГО РЕШЕНИЯ, ПРОЯВИТЕ СВОЮ ФАНТАЗИЮ!20.25 Распределения
Выберите любое непрерывное распределение из представленных в базовом пакете stats или же в любом другом пакете. Найти все распределения пакета stats можно с помощью ?Distributions. Подберите для него какие-нибудь параметры или используйте параметры по умолчанию.
Я возьму F-распределение с параметрами
df1 = 4иdf = 10, но вы можете выбрать другое распределение.
- Визуализируйте функцию плотности вероятности для выбранного распределения.
v <- seq(0, 5, 0.01)
plot(v, df(v, df1 = 4, df2 = 10))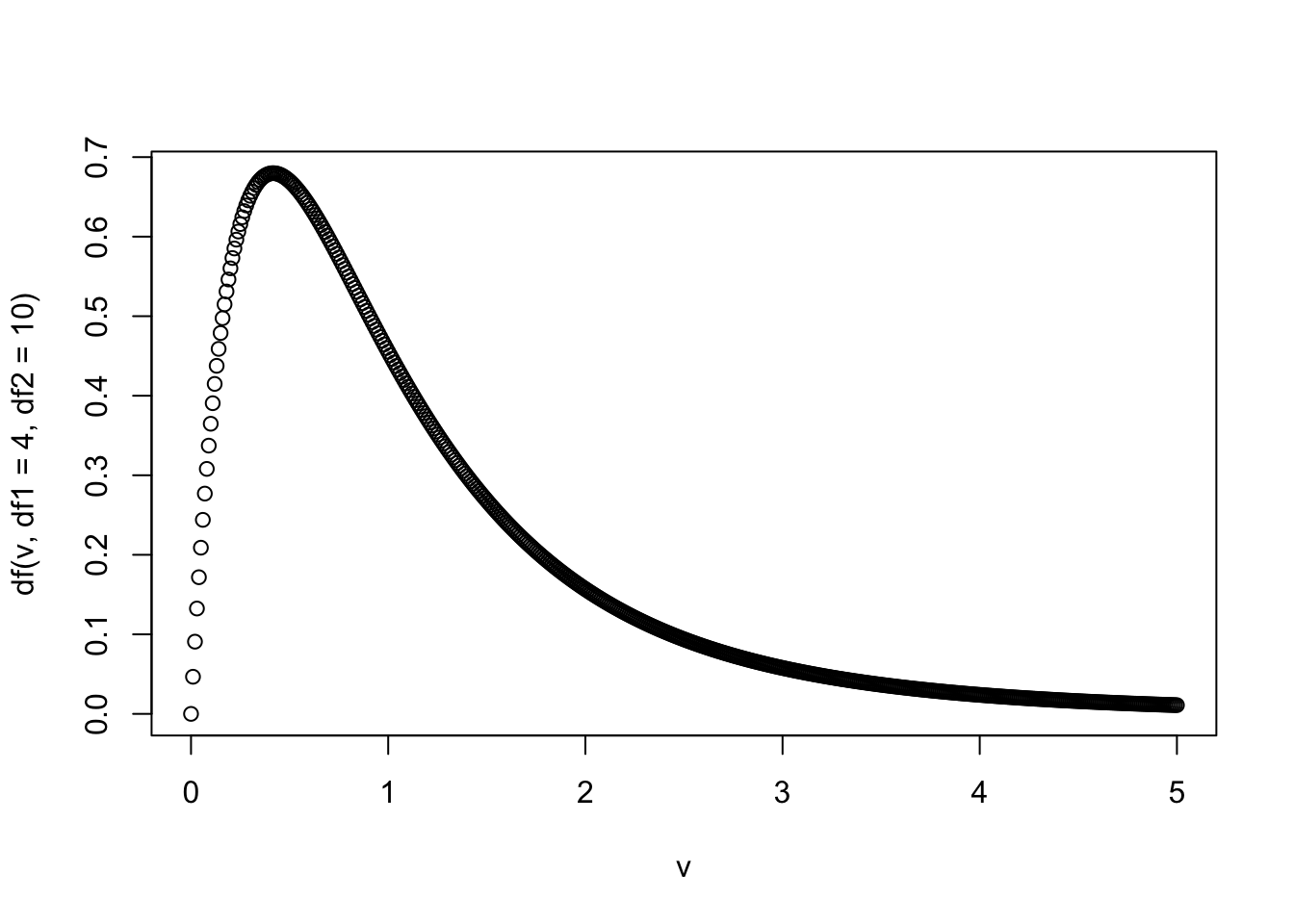
- Визуализируйте функцию накопленной плотности распределения для выбранной функции.
plot(v, pf(v, df1 = 4, df2 = 10))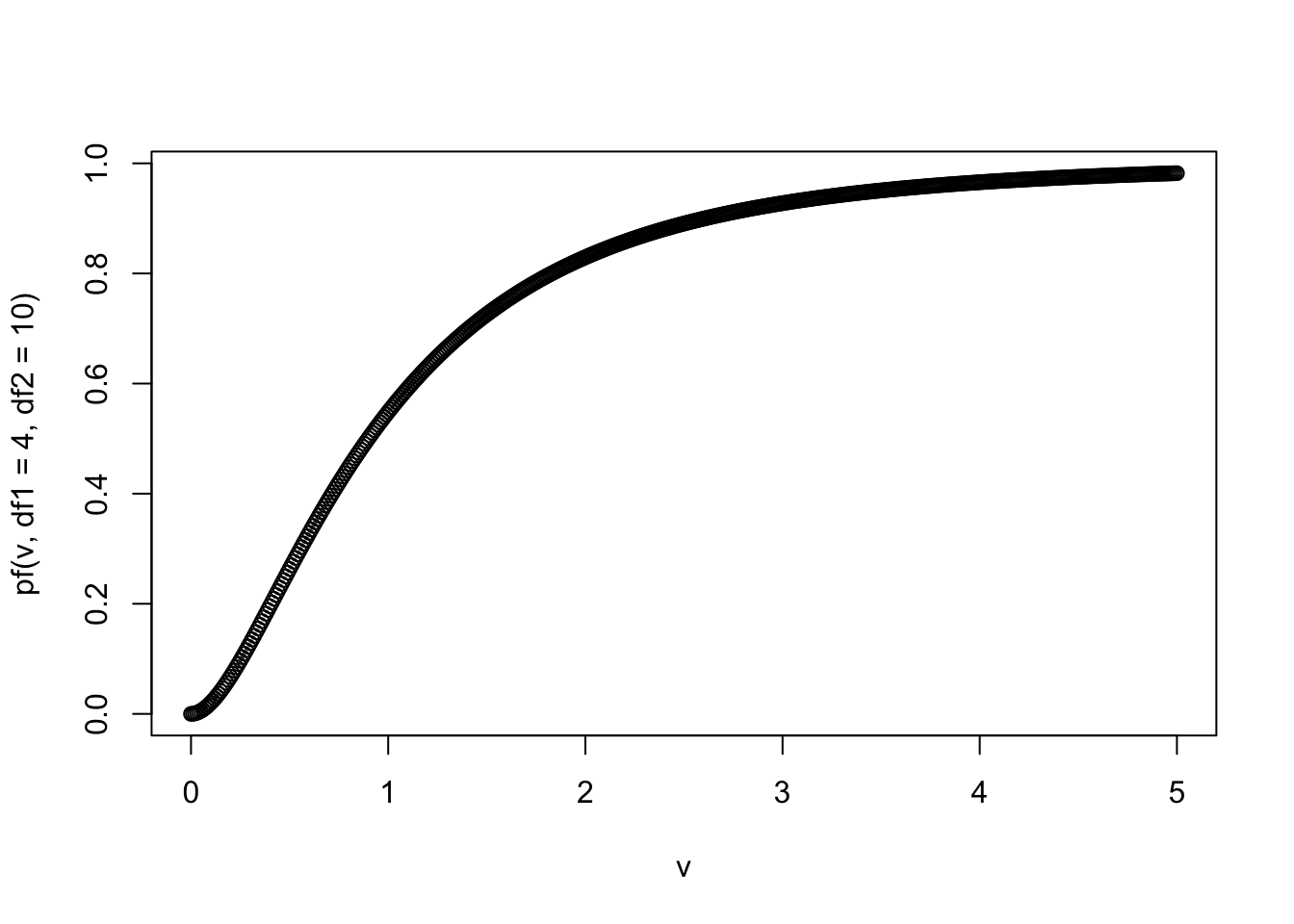
- Визуализируйте квантильную функцию для выбранного распределения.
p <- seq(0, 1, .01)
plot(p, qf(p, df1 = 4, df2 = 10))
- Сделайте выборку из 100 случайных значений из выбранного распределения и постройте гистограмму (функция
hist()) для полученной выборки.
hist(rf(100, df1 = 4, df2 = 10))
20.26 Одновыборочный t-test
- Представьте, что наши супергерои из набора данных
heroes— это выборка из генеральной совокупности всех написанных и ненаписанных супергероев. Проведите одновыборочный t-тест для веса супергероев и числа 100 — предположительного среднего веса в генеральной совокупности всех супергероев. Проинтерпретируйте результат.
t.test(heroes$Weight, mu = 100)##
## One Sample t-test
##
## data: heroes$Weight
## t = 2.6174, df = 494, p-value = 0.009133
## alternative hypothesis: true mean is not equal to 100
## 95 percent confidence interval:
## 103.0549 121.4501
## sample estimates:
## mean of x
## 112.2525p-value меньше .05, поэтому мы можем отклонить нулевую гипотезу о том, что среднее для веса в генеральной совкупности, из которой вы взяли выборку супергероев, равно 100. Мы принимаем ненулевую гипотезу о том, что в генеральной совокупности средний вес не равен 100.
- Проведите одновыборочный t-тест для роста супергероев и числа 185 — предположительного среднего роста в генеральной совокупности всех супергероев. Проинтерпретируйте результат.
t.test(heroes$Height, mu = 185)##
## One Sample t-test
##
## data: heroes$Height
## t = 0.66246, df = 516, p-value = 0.508
## alternative hypothesis: true mean is not equal to 185
## 95 percent confidence interval:
## 181.6068 191.8458
## sample estimates:
## mean of x
## 186.7263p-value больше .05, поэтому мы не можем отклонить нулевую гипотезу о том, что среднее для роста в генеральной совкупности, из которой вы взяли выборку супергероев, равно 185.
20.27 Двухвыборочный зависимый t-test
diet <- readr::read_csv("https://raw.githubusercontent.com/Pozdniakov/tidy_stats/master/data/stcp-Rdataset-Diet.csv")- Посчитайте двухвыборочный зависимый т-тест для остальных диет: для диеты 2 и диеты 3. Проинтерпретируйте полученные результаты.
diet2 <- diet %>%
filter(Diet == 2)
t.test(diet2$pre.weight, diet2$weight6weeks, paired = TRUE)##
## Paired t-test
##
## data: diet2$pre.weight and diet2$weight6weeks
## t = 6.231, df = 26, p-value = 1.36e-06
## alternative hypothesis: true difference in means is not equal to 0
## 95 percent confidence interval:
## 2.027715 4.024137
## sample estimates:
## mean of the differences
## 3.025926p-value меньше .05, поэтому мы можем отклонить нулевую гипотезу о том, что масса до и после диеты одинаковая. Мы принимаем альтернативную гипотезу о различии средних в генеральной совокупности.
diet3 <- diet %>%
filter(Diet == 3)
t.test(diet3$pre.weight, diet3$weight6weeks, paired = TRUE)##
## Paired t-test
##
## data: diet3$pre.weight and diet3$weight6weeks
## t = 11.167, df = 26, p-value = 2.03e-11
## alternative hypothesis: true difference in means is not equal to 0
## 95 percent confidence interval:
## 4.200493 6.095803
## sample estimates:
## mean of the differences
## 5.148148p-value меньше .05, поэтому мы можем отклонить нулевую гипотезу о том, что масса до и после диеты одинаковая. Мы принимаем альтернативную гипотезу о различии средних в генеральной совокупности.
20.28 Двухвыборочный независимый t-test
- Сделайте независимый t-тест для сравнения веса испытуемых двух групп после диеты, сравнив вторую и третью группу. Проинтерпретируйте результаты.
diet23 <- diet %>%
filter(Diet %in% 2:3)
t.test(weight6weeks ~ Diet, data = diet23, paired = FALSE)##
## Welch Two Sample t-test
##
## data: weight6weeks by Diet
## t = -0.15686, df = 49.774, p-value = 0.876
## alternative hypothesis: true difference in means is not equal to 0
## 95 percent confidence interval:
## -5.471327 4.678734
## sample estimates:
## mean in group 2 mean in group 3
## 68.08519 68.48148p-value больше .05, поэтому мы не можем отклонить нулевую гипотезу о том, что масса до и после диеты одинаковая.
- Сделайте независимый t-тест для сравнения веса испытуемых двух групп после диеты, сравнив первую и третью группу. Проинтерпретируйте результаты.
diet13 <- diet %>%
filter(Diet %in% c(1,3))
t.test(weight6weeks ~ Diet, data = diet13, paired = FALSE)##
## Welch Two Sample t-test
##
## data: weight6weeks by Diet
## t = 0.46818, df = 48.072, p-value = 0.6418
## alternative hypothesis: true difference in means is not equal to 0
## 95 percent confidence interval:
## -3.602450 5.789487
## sample estimates:
## mean in group 1 mean in group 3
## 69.57500 68.48148p-value больше .05, поэтому мы не можем отклонить нулевую гипотезу о том, что масса до и после диеты одинаковая.
20.29 Непараметрические аналоги t-теста
- Сравните вес первой и второй группы после диеты, используя тест Манна-Уитни. Сравните результаты теста Манна-Уитни с результатами t-теста? Проинтерпретируйте полученные результаты.
diet12 <- diet %>%
filter(Diet %in% c(1,2))
wilcox.test(weight6weeks ~ Diet, data = diet12, paired = FALSE)##
## Wilcoxon rank sum test with continuity correction
##
## data: weight6weeks by Diet
## W = 368, p-value = 0.4117
## alternative hypothesis: true location shift is not equal to 0В обоих случаях p-value больше 0.05, мы не можем отклонить нулевую гипотезу об отсутствии различий.
- Повторите задание для второй и третьей группы, а так же для первой и третьей группы.
wilcox.test(weight6weeks ~ Diet, data = diet23, paired = FALSE)##
## Wilcoxon rank sum test with continuity correction
##
## data: weight6weeks by Diet
## W = 340.5, p-value = 0.6843
## alternative hypothesis: true location shift is not equal to 0wilcox.test(weight6weeks ~ Diet, data = diet13, paired = FALSE)##
## Wilcoxon rank sum test with continuity correction
##
## data: weight6weeks by Diet
## W = 346, p-value = 0.6849
## alternative hypothesis: true location shift is not equal to 0В обоих случаях p-value больше 0.05, как и для соответствующих t-тестов. Мы не можем отклонить нулевую гипотезу об отстутствии различий между второй и третьей диетой, между первой и третьей диетой.
- Сравните вес до и после для диеты 1, используя тест Уилкоксона. Сравните с результатами применения t-теста. Проинтерпретируйте полученные результаты.
diet1 <- diet %>%
filter(Diet == 1)
wilcox.test(diet1$pre.weight, diet1$weight6weeks, paired = TRUE)##
## Wilcoxon signed rank test with continuity correction
##
## data: diet1$pre.weight and diet1$weight6weeks
## V = 299, p-value = 2.203e-05
## alternative hypothesis: true location shift is not equal to 0И t-тест, и тест Уилкоксона дают p-value ниже 0.05. Мы можем отклонить нулевую гипотезу об отсутствии различий.
- Сравните вес до и после для диеты 2 и диеты 3, используя тест Уилкоксона. Сравните с результатами применения t-теста. Проинтерпретируйте полученные результаты.
wilcox.test(diet2$pre.weight, diet2$weight6weeks, paired = TRUE)##
## Wilcoxon signed rank test with continuity correction
##
## data: diet2$pre.weight and diet2$weight6weeks
## V = 313, p-value = 5.419e-05
## alternative hypothesis: true location shift is not equal to 0wilcox.test(diet3$pre.weight, diet3$weight6weeks, paired = TRUE)##
## Wilcoxon signed rank test with continuity correction
##
## data: diet3$pre.weight and diet3$weight6weeks
## V = 378, p-value = 5.912e-06
## alternative hypothesis: true location shift is not equal to 0В обоих случаях и t-тест, и тест Уилкоксона дают p-value ниже 0.05. Мы можем отклонить нулевую гипотезу об отсутствии различий.
20.30 Исследование набора данных Backpack
Для следующих тем нам понадобится набор данных Backpack из пакета Stat2Data.
#install.packages("Stat2Data")
library(Stat2Data)
data(Backpack)
back <- Backpack %>%
mutate(backpack_kg = 0.45359237 * BackpackWeight,
body_kg = 0.45359237 * BodyWeight)- Как различается вес рюкзака в зависимости от пола? Кто весит больше?
back %>%
group_by(Sex) %>%
summarise(mean(backpack_kg))- Если допустить, что выборка репрезентативна, то можно ли сделать вывод о различии по среднему весу рюкзаков в генеральной совокупности?
t.test(backpack_kg ~ Sex, data = back)##
## Welch Two Sample t-test
##
## data: backpack_kg by Sex
## t = -1.1782, df = 86.25, p-value = 0.242
## alternative hypothesis: true difference in means is not equal to 0
## 95 percent confidence interval:
## -1.6892365 0.4320067
## sample estimates:
## mean in group Female mean in group Male
## 5.006010 5.634625p-value больше .05, поэтому мы не можем отклонить нулевую гипотезу о том, что масса до и после диеты одинаковая.
- Повторите пунктs 2 и 3 для веса самих студентов.
back %>%
group_by(Sex) %>%
summarise(mean(body_kg))t.test(body_kg ~ Sex, data = back)##
## Welch Two Sample t-test
##
## data: body_kg by Sex
## t = -7.0863, df = 77.002, p-value = 5.704e-10
## alternative hypothesis: true difference in means is not equal to 0
## 95 percent confidence interval:
## -20.32511 -11.40803
## sample estimates:
## mean in group Female mean in group Male
## 62.28236 78.14893p-value меньше .05, поэтому мы можем отклонить нулевую гипотезу о том, что масса студентов-мужчин и студентов-женщин одинаковая. Мы принимаем альтернативную гипотезу о различии средних в генеральной совокупности.
- Визуализируйте распределение этих двух переменных в зависимости от пола (используя
ggplot2)
library(ggplot2)
ggplot(back)+
geom_histogram(aes(x = body_kg, fill = Sex), bins = 15, position = "identity", alpha = 0.7)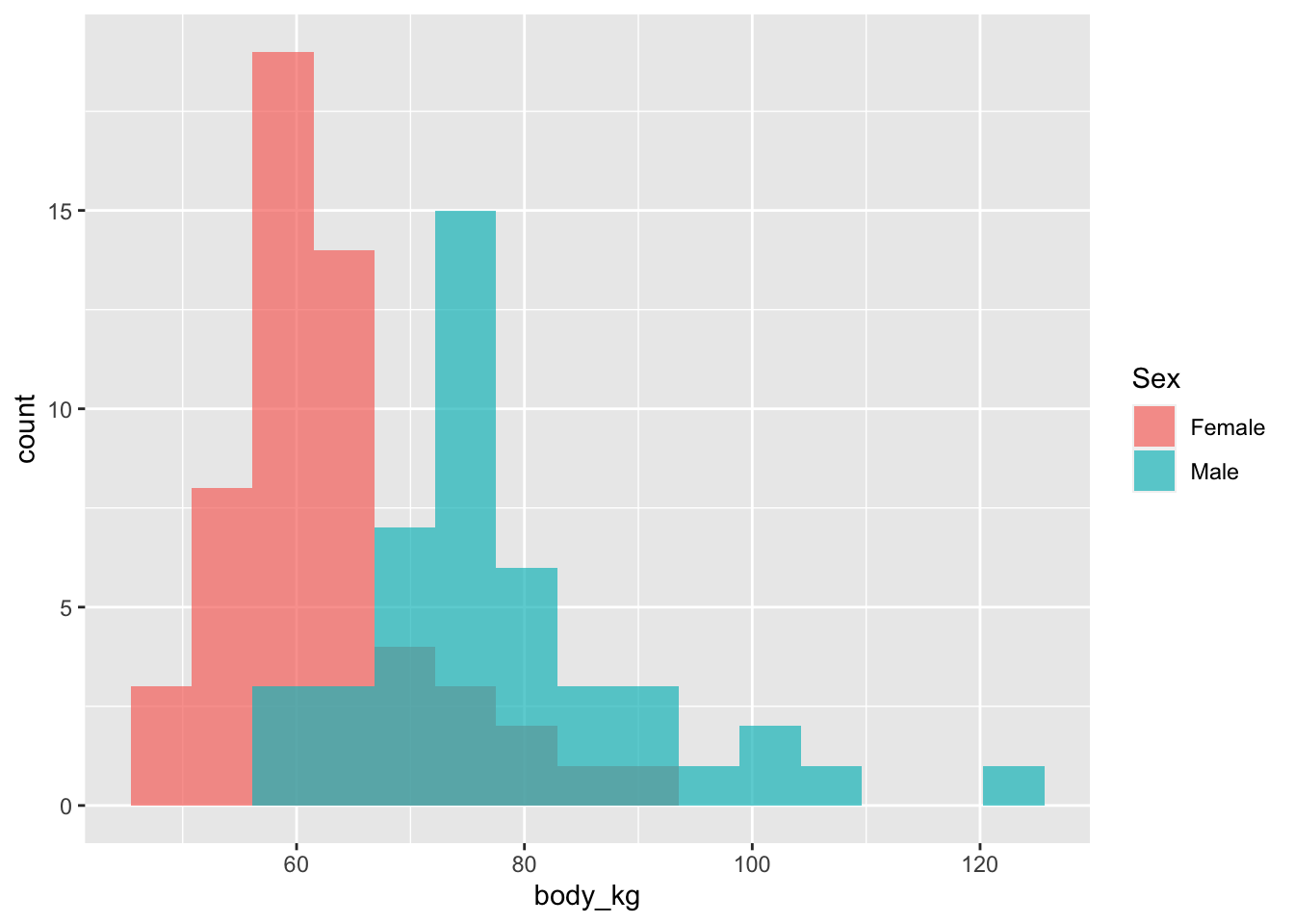
- Постройте диаграмму рассеяния с помощью
ggplot2. Цветом закодируйте пол респондента.
ggplot(back, aes(x = body_kg, y = backpack_kg))+
geom_point(aes(colour = Sex), alpha = 0.5, size = 2)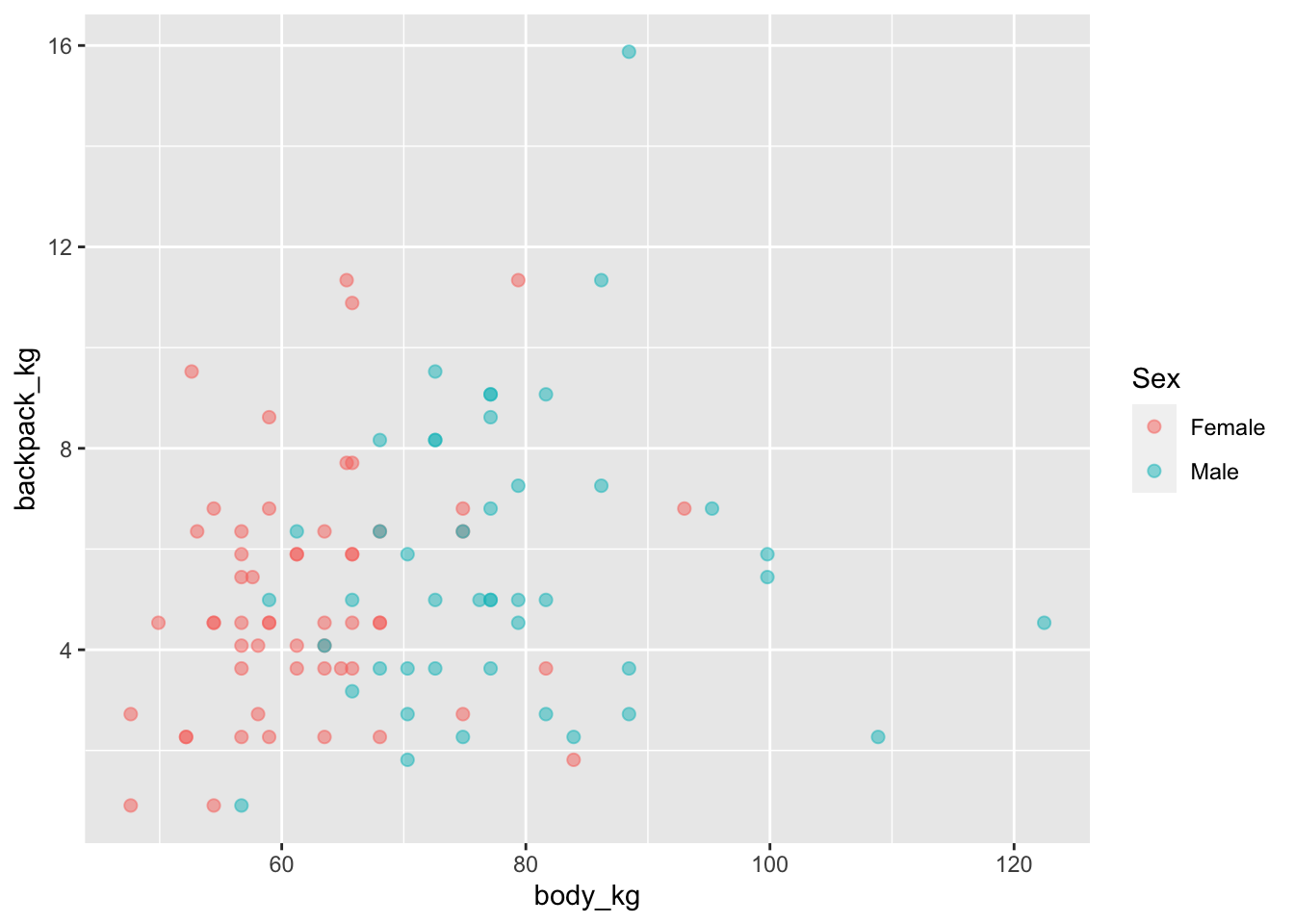
20.31 Ковариация
- Посчитайте матрицу ковариаций для веса студентов и их рюкзаков в фунтах. Различаются ли результаты подсчета ковариации этих двух переменных от результатов подсчета ковариаций веса студентов и их рюкзаков в килограммах? Почему?
back %>%
select(BodyWeight, BackpackWeight) %>%
cov()## BodyWeight BackpackWeight
## BodyWeight 864.20960 32.08788
## BackpackWeight 32.08788 33.23677Результаты различаются, потому что значение ковариации зависит от размерности исходных шкал.
20.32 Коэффициент корреляции
- Посчитайте коэффициент корреляции Пирсона для веса студентов и их рюкзаков в фунтах. Различаются ли результаты подсчета коэффициента корреляции Пирсона (сам коэффициент, p-value) этих двух переменных от результатов подсчета корреляции Пирсона веса студентов и их рюкзаков в килограммах? Почему?
cor.test(back$BackpackWeight, back$BodyWeight)##
## Pearson's product-moment correlation
##
## data: back$BackpackWeight and back$BodyWeight
## t = 1.9088, df = 98, p-value = 0.05921
## alternative hypothesis: true correlation is not equal to 0
## 95 percent confidence interval:
## -0.007360697 0.371918344
## sample estimates:
## cor
## 0.1893312Результаты не различаются, потому что значение ковариации не зависит от размерности исходных шкал.
- Посчитайте коэффициент корреляции Пирсона для веса и роста супергероев из датасета
heroes. Проинтерпретируйте результат.
cor.test(heroes$Weight, heroes$Height)##
## Pearson's product-moment correlation
##
## data: heroes$Weight and heroes$Height
## t = 4.3555, df = 488, p-value = 1.619e-05
## alternative hypothesis: true correlation is not equal to 0
## 95 percent confidence interval:
## 0.1066877 0.2772717
## sample estimates:
## cor
## 0.1934412p-value меньше 0.05, поэтому мы можем отклонить нулевую гипотезу об отсутствии линейной связи между ростом и весом и принять альтернативную гипотезу о том, что такая связь есть. Судя по знаку и размеру корреляции, чем выше рост, тем выше вес супергероя, но эта связь достаточно слабая.
- Теперь посчитайте коэффициент корреляции Спирмена и коэффициент корреляции Кэнделла для веса и роста супергероев из датасета
heroes. Различаются ли результаты по сравнению с коэффициентом корреляции Пирсона? Почему?
cor.test(heroes$Weight, heroes$Height, method = "spearman")##
## Spearman's rank correlation rho
##
## data: heroes$Weight and heroes$Height
## S = 3915061, p-value < 2.2e-16
## alternative hypothesis: true rho is not equal to 0
## sample estimates:
## rho
## 0.8003344cor.test(heroes$Weight, heroes$Height, method = "kendall")##
## Kendall's rank correlation tau
##
## data: heroes$Weight and heroes$Height
## z = 21.548, p-value < 2.2e-16
## alternative hypothesis: true tau is not equal to 0
## sample estimates:
## tau
## 0.6751664В обоих случаях p-value меньше 0.05, поэтому мы можем отклонить нулевую гипотезу об отсутствии связи между ростом и весом и принять альтернативную гипотезу о том, что такая связь есть. Сильное различие между коэффициентами корреляции указывает на нелинейность этой связи, либо же на наличие значительных выбросов в данных.