9 Продвинутый tidyverse
9.1 Трансформация нескольких колонок: dplyr::across()
Допустим, вы хотите посчитать среднюю массу и рост, группируя по полу супергероев. Можно посчитать это внутри одного summarise(), использую запятую:
heroes %>%
group_by(Gender) %>%
summarise(height = mean(Height, na.rm = TRUE),
weight = mean(Weight, na.rm = TRUE))Если таких колонок будет много, то это уже станет сильно неудобным, нам придется много копировать код, а это чревато ошибками и очень скучно.
Поэтому в dplyr есть функция для операций над несколькими колонками сразу: dplyr::across()26. Эта функция работает похожим образом на функции семейства apply() и использует tidyselect для выбора колонок.
Таким образом, конструкции с функцией across() можно разбить на три части:
- Выбор колонок с помощью tidyselect. Здесь работают все те приемы, которые мы изучили при выборе колонок (8.10.2).
- Собственно применение функции
across(). Первый аргумент.col— колонки, выбранные на первом этапе с помощью tidyselect, по умолчанию этоeverything(), т.е. все колонки. Второй аргумент.fns— это функция или целый список из функций, которые будут применены к выбранным колонкам. Если функции требуют дополнительных аргументов, то они могут быть перечислены внутриacross(). - Использование
summarise()или другой функцииdplyr. В этом случае в качестве аргумента для функции используется результат работы функцииacross().
Вот такой вот бутерброд выходит. Давайте посмотрим, как это работает на практике и посчитаем среднее значение по колонкам Height и Weight.
heroes %>%
group_by(Gender) %>%
summarise(across(c(Height,Weight), mean))Здесь мы столкнулись с уже известной нам проблемой: функция mean() при столкновении хотя бы с одним NA будет возвращать NA, если мы не изменим параметр na.rm =. Как и в случае с функциями семейства apply() (@ref(apply_f)), дополнительные параметры для функции можно перечислить через запятую после самой функции:
heroes %>%
group_by(Gender) %>%
summarise(across(c(Height, Weight), mean, na.rm = TRUE))До этого мы просто использовали выбор колонок по их названию. Но именно внутри across() использование tidyselect раскрывается как удивительно элегантный и мощный инструмент. Например, можно посчитать среднее для всех numeric колонок:
heroes %>%
drop_na(Height, Weight) %>%
group_by(Gender) %>%
summarise(across(where(is.numeric), mean, na.rm = TRUE))Или длину строк для строковых колонок. Для этого нам понадобится вспомнить, как создавать анонимные функции (@ref(anon_f)).
heroes %>%
group_by(Gender) %>%
summarise(across(where(is.character),
function(x) mean(nchar(x), na.rm = TRUE)))Или же даже посчитать и то, и другое внутри одного summarise()!
heroes %>%
group_by(Gender) %>%
summarise(across(where(is.numeric), mean, na.rm = TRUE),
across(where(is.character),
function(x) mean(nchar(x), na.rm = TRUE)))Внутри одного across() можно применить не одну функцию к каждой из выбранных колонок, а сразу несколько функций для каждой из колонок. Для этого нам нужно использовать список функций (желательно - проименованный).
heroes %>%
group_by(Gender) %>%
summarise(across(c(Height, Weight),
list(minimum = min,
average = mean,
maximum = max),
na.rm = TRUE))Вот нам и понадобился список функций (@ref(functions_objects))!
heroes %>%
group_by(Gender) %>%
summarise(across(c(Height, Weight),
list(min = function(x) min(x, na.rm = TRUE),
mean = function(x) mean(x, na.rm = TRUE),
max = function(x) max(x, na.rm = TRUE),
na_n = function(x, ...) sum(is.na(x)))
)
)Хотя основное применение функции across() — это массовое подытоживание с помощью summarise(), across() можно использовать и с другими функциями dplyr. Например, можно делать массовые операции с колонками с помощью mutate():
heroes %>%
mutate(across(where(is.character), as.factor))Менее очевидный способ применения across() - использование across() внутри count() вместе с функцией n_distinct(), которая считает количество уникальных значений в векторе. Это позволяет посмотреть таблицу частот для группирующих переменных:
heroes %>%
select(where(function(x) n_distinct(x) <= 6))heroes %>%
count(across(where(function(x) n_distinct(x) <= 6)))9.2 Объединение нескольких датафреймов
9.2.1 Соединение структурно схожих датафреймов: bind_rows(), bind_cols()
Для начала создадим следующие тибблы и сохраним их как dc, marvel и other_publishers:
dc <- heroes %>%
filter(Publisher == "DC Comics") %>%
group_by(Gender) %>%
summarise(weight_mean = mean(Weight, na.rm = TRUE))
dcmarvel <- heroes %>%
filter(Publisher == "Marvel Comics") %>%
group_by(Gender) %>%
summarise(weight_mean = mean(Weight, na.rm = TRUE))
marvelother_publishers <- heroes %>%
filter(!(Publisher %in% c("DC Comics","Marvel Comics"))) %>%
group_by(Gender) %>%
summarise(weight_mean = mean(Weight, na.rm = TRUE))
other_publishersНесколько тибблов можно объединить вертикально с помощью функции bind_rows(). Для корректного объединения тибблы должны иметь одинаковые названия колонок.
bind_rows(dc, marvel)Чтобы соединить тибблы горизонтально, воспользуйтесь функцией bind_cols().
bind_cols(dc, marvel)## New names:
## * Gender -> Gender...1
## * weight_mean -> weight_mean...2
## * Gender -> Gender...3
## * weight_mean -> weight_mean...4Функции bind_rows() и bind_cols() могут работать не только с двумя, но сразу с несколькими датафреймами.
bind_rows(dc, marvel, other_publishers)На входе в функции bind_rows() и bind_cold() можно подавать как сами датафреймы или тибблы через запятую, так и список из датафреймов/тибблов.
heroes_list_of_df <- list(DC = dc,
Marvel = marvel,
Other = other_publishers)
bind_rows(heroes_list_of_df)Чтобы не потерять, из какого датафрейма какие данные, можно указать любое строковое значение (название будущей колонки) для необязательного аргумента .id =.
bind_rows(heroes_list_of_df, .id = "Publisher")bind_rows() обычно используется, когда ваши данные находятся в разных файлах с одинаковой структурой. Тогда вы можете прочитать все таблицы в папке, сохранить их в качестве списка из датафреймов и объединить в один датафрейм с помощью bind_rows().
9.2.2 Реляционные данные: *_join()
В реальности иногда возникает ситуация, когда нужно соединить две таблички, у которых есть общий столбец (или несколько столбцов), но все остальные столбцы различаются. Табличек может быть и больше, это может быть целая сеть таблиц, некоторые из которых содержат основные данные, а некоторые - дополнительные, которые необходимо на определенном этапе “включить” в анализ. Например, таблица с расшифровкой аббревиатур или сокращений вроде коротких названий стран или таблица телефонных кодов разных стран. Совокупность нескольких связанных друг с другом таблиц называют реляционными данными.
В случае с реляционными данными простых bind_rows() и bind_cols() становится недостаточно.
Эти две таблички нужно объединить (join). Эта задача обычно возникает не очень часто, обычно это происходит один-два раза в одном проекте, когда нужно дополнить имеющиеся данные дополнительной информацией извне или объединить два набора данных, обрабатывавшихся в разных программах. Всякий раз, когда такая задача возникает, это доставляет много боли. dplyr предлагает интуитивно понятный инструмент для объединения реляционных данных - семейство функций *_join().
Возьмем для примера два тиббла band_members и band_instruments, встроенных в dplyr специально для демонстрации работы функций *_join().
band_membersband_instrumentsУ этих двух тибблов есть колонка с одинаковым названием, которая по своему смыслу соединяет данные обоих тибблов. Такая колонка называется ключом. Ключ должен однозначно идентифицировать наблюдения27.
Давайте попробуем посоединять band_members и band_instruments разными вариантами *_join() и посмотрим, что у нас получится. Все эти функции имеют на входе два обязательных аргумента (x = и y =) в которые мы должны подставить два датафрейма/тиббла которые мы хотим объединить. Главное различие между этими функциями заключается в том, что они будут делать, если уникальные значения в ключах x и y не соответствуют друг другу.
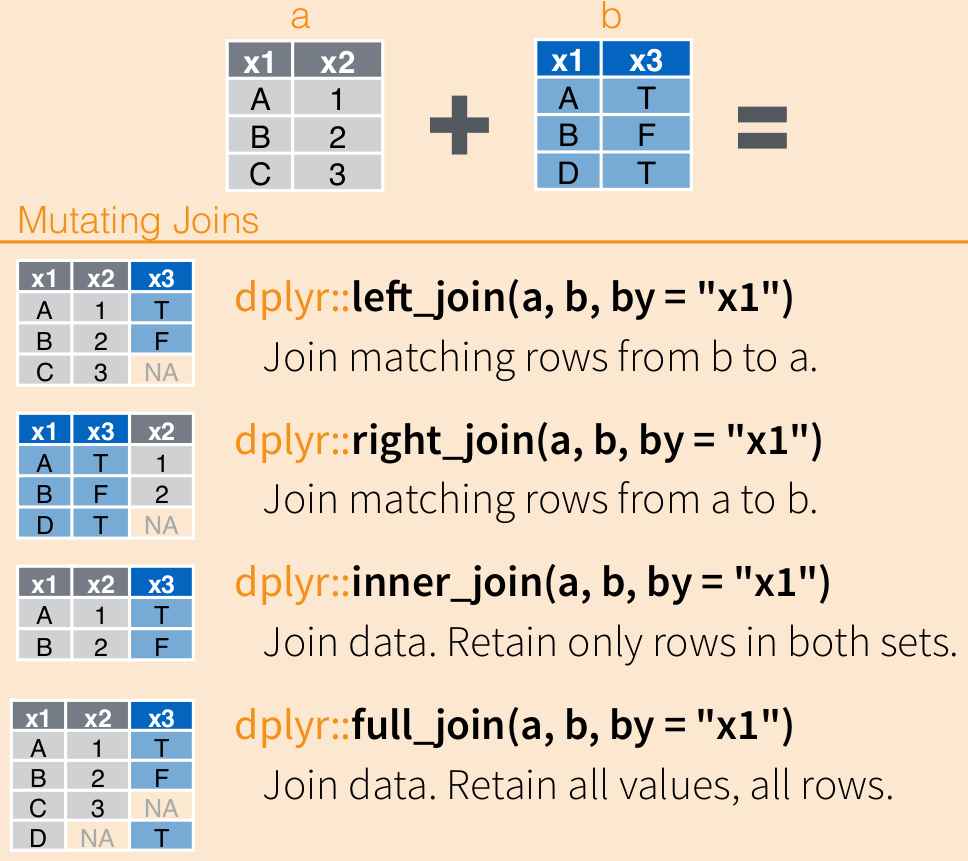
left_join():
band_members %>%
left_join(band_instruments)## Joining, by = "name"left_join() - это самая простая для понимания и самая используемая функция из семейства *_join(). Она как бы “дополняет” информацию из первого тиббла вторым тибблом. В этом случае сохраняются все уникальные наблюдения в x, но отбрасываются лишние наблюдения в тиббле y. Тем значениям, которым не нашлось соотвествия в y, в колонках, взятых их y, ставятся значения NA.
Вы можете сами задать колонки-ключи параметром by =, по умолчанию это все колонки с одинаковыми названиями в двух тибблах.
band_members %>%
left_join(band_instruments, by = "name")Часто случается, что колонки-ключи называются по-разному в двух тибблах. Их необязательно переименовывать, можно поставить соответстие вручную используя проименованный вектор:
band_members %>%
left_join(band_instruments2, by = c("name" = "artist"))right_join():
band_members %>%
right_join(band_instruments)## Joining, by = "name"right_join() отбрасывает строчки в x, которых не было в y, но сохраняет соответствующие строчки y - left_join() наоборот.
full_join():
band_members %>%
full_join(band_instruments)## Joining, by = "name"Функция full_join() сохраняет все строчки и из x и y. Пожалуй, наиболее используемая функция после left_join() — благодаря full_join() вы точно ничего не потеряете при объединении.
inner_join():
band_members %>%
inner_join(band_instruments)## Joining, by = "name"Функция full_join() сохраняет только строчки, которые присутствуют и в x, и в y.
semi_join():
band_members %>%
semi_join(band_instruments)## Joining, by = "name"anti_join():
band_members %>%
anti_join(band_instruments)## Joining, by = "name"Функции semi_join() и anti_join() не присоединяют второй датафрейм/тиббл (y) к первому. Вместо этого они используются как некоторый словарь-фильтр для отделения только тех значений в x, которые есть в y (semi_join()) или, наоборот, которых нет в y (anti_join()).
9.3 Tidy data: tidyr::pivot_longer(), tidyr::pivot_wider()
Принцип tidy data предполагает, что каждая строчка содержит в себе одно измерение, а каждая колонка - одну характеристику. Тем не менее, это не говорит однозначно о том, как именно хранить повторные измерения. Их можно хранить как одну колонку для каждого измерения (широкий формат) и как две колонки: одна колонка - для идентификатора измерения, другая колонка - для записи самого измерения.
Это лучше понять на примере. Например, вес до и после прохождения курса. Как это лучше записать - как два числовых столбца (один испытуемый - одна строка) или же создать отдельную “группирующую” колонку, в которой будет написано время измерения, а в другой - измеренные значения (одно измерение - одна строка)?
- Широкий формат:
| Студент | До курса по R | После курса по R |
|---|---|---|
| Маша | 70 | 63 |
| Рома | 80 | 74 |
| Антонина | 86 | 71 |
- Длинный" формат:
| Студент | Время измерения | Масса (кг) |
|---|---|---|
| Маша | До курса по R | 70 |
| Рома | До курса по R | 80 |
| Антонина | До курса по R | 86 |
| Маша | После курса по R | 63 |
| Рома | После курса по R | 74 |
| Антонина | После курса по R | 71 |
На самом деле, оба варианта приемлимы, оба варианта возможны в реальных данных, а разные функции и статистические пакеты могут требовать от вас как длинный, так и широкий форматы.
Таким образом, нам нужно научиться переводить из широкого формата в длинный и наоборот.
tidyr::pivot_longer(): из широкого в длинный форматtidyr::pivot_wider(): из длинного в широкий формат

new_diet <- tibble(
student = c("Маша", "Рома", "Антонина"),
before_r_course = c(70, 80, 86),
after_r_course = c(63, 74, 71)
)
new_dietТиббл new_diet - это пример широкого формата данных.
Превратим тиббл new_diet длинный:
new_diet %>%
pivot_longer(cols = before_r_course:after_r_course,
names_to = "measurement_time",
values_to = "weight_kg")А теперь обратно в короткий:
new_diet %>%
pivot_longer(cols = before_r_course:after_r_course,
names_to = "measurement_time",
values_to = "weight_kg") %>%
pivot_wider(names_from = "measurement_time",
values_from = "weight_kg")9.4 Функциональное программирование: purrr
purrr — это пакет для функционального программирования в tidyverse. Как и многие пакеты tidyverse, purrr пытается заменить собой базовый функционал R на более понятный и удобный. В данном случае, речь в первую очередь идет о функциях семейства apply(), с которыми мы работали ранее (@ref(apply_f)).
Давайте вспомним, как работает lapply(). В качестве первого аргумента функция lapply() принимает список (или то, что может быть в него превращено, например, датафрейм), в качестве второго - функцию, которая будет применена к каждому элементу списка. На выходе мы получим список такой же длины.
lapply(heroes, class)## $X1
## [1] "numeric"
##
## $name
## [1] "character"
##
## $Gender
## [1] "character"
##
## $`Eye color`
## [1] "character"
##
## $Race
## [1] "character"
##
## $`Hair color`
## [1] "character"
##
## $Height
## [1] "numeric"
##
## $Publisher
## [1] "character"
##
## $`Skin color`
## [1] "character"
##
## $Alignment
## [1] "character"
##
## $Weight
## [1] "numeric"Функция purrr::map() работает по тому же принципу: можно просто заменить lapply() на map(), и мы получим тот же результат.
map(heroes, class)## $X1
## [1] "numeric"
##
## $name
## [1] "character"
##
## $Gender
## [1] "character"
##
## $`Eye color`
## [1] "character"
##
## $Race
## [1] "character"
##
## $`Hair color`
## [1] "character"
##
## $Height
## [1] "numeric"
##
## $Publisher
## [1] "character"
##
## $`Skin color`
## [1] "character"
##
## $Alignment
## [1] "character"
##
## $Weight
## [1] "numeric"map() можно встроить в канал с пайпом (впрочем, как и lapply()):
heroes %>%
map(class)## $X1
## [1] "numeric"
##
## $name
## [1] "character"
##
## $Gender
## [1] "character"
##
## $`Eye color`
## [1] "character"
##
## $Race
## [1] "character"
##
## $`Hair color`
## [1] "character"
##
## $Height
## [1] "numeric"
##
## $Publisher
## [1] "character"
##
## $`Skin color`
## [1] "character"
##
## $Alignment
## [1] "character"
##
## $Weight
## [1] "numeric"Как и lapply(), map() всегда возвращает список. Из-за этого мы больше пользовались функцией sapply(), а не lapply(). Функция sapply() упрощала результат до вектора, если это возможно. Подобное упрощение может показаться удобным пока не сталкиваешься с тем, что иногда очень сложно предсказать, какой тип данных получится на выходе. Есть функция vapply() в которой можно управлять типом данных на выходе, но она не очень удобная. В purrr эта проблема решена просто: есть множество функций map_*(), где вместо звездочки - нужный формат на выходе.
Например, если мы хотим получить строковый вектор на выходе, то нам нужна функция map_chr().
heroes %>%
map_chr(class)## X1 name Gender Eye color Race Hair color
## "numeric" "character" "character" "character" "character" "character"
## Height Publisher Skin color Alignment Weight
## "numeric" "character" "character" "character" "numeric"Можно превратить результат в датафрейм с помощью map_df().
heroes %>%
map_df(class)Так же как и функции семейства apply(), функции map_*() отлично сочетаются с анонимными функциями.
heroes %>%
map_int(function(x) sum(is.na(x)))## X1 name Gender Eye color Race Hair color Height
## 0 0 29 172 304 172 217
## Publisher Skin color Alignment Weight
## 0 662 7 239Однако у purrr есть свой, более короткий способ записи анонимных функций: function(arg) заменяется на ~, а arg на ..
heroes %>%
map_int(~sum(is.na(.)))## X1 name Gender Eye color Race Hair color Height
## 0 0 29 172 304 172 217
## Publisher Skin color Alignment Weight
## 0 662 7 239Если нужно итерироваться сразу по нескольким спискам, то есть функции map2_*() (для двух списков) и pmap_*() (для нескольких списков).
9.5 Колонки-списки и нестинг: nest()
Ранее мы говорили о том, что датафрейм — это по своей сути список из векторов разной длины. На самом деле, это не совсем так: колонки обычного датафрейма вполне могут быть списками. Однако делать так обычно не рекомендуется, пусть R это и не запрещает создавать такие колонки: многие функции предполагают, что все колонки датафрейма являются векторами.
tidyverse гораздо дружелюбнее относится к использовании списка в качестве колонки. Такие колонки называются колонками-списками (list columns). Основной способ их создания - использования функции tidyr::nest(). С помощью tidyselect нужно выбрать сжимаемые колонки, которые будут агрегированы по невыбранным колонками. Это и называется нестингом.
heroes %>%
nest(!Gender)## Warning: All elements of `...` must be named.
## Did you want `data = c(X1, name, `Eye color`, Race, `Hair color`, Height, Publisher,
## `Skin color`, Alignment, Weight)`?Заметьте, у нас появилась колонка data, в которой содержатся тибблы. Туда и спрятались все наши данные.
Нестинг похож на агрегирование с помощью group_by(). Если сделать нестинг сгруппированного с помощью group_by() тиббла, то сожмутся все колонки кроме тех, которые выступают в качестве групп:
heroes %>%
group_by(Gender) %>%
nest()Теперь можно работать с колонкой-списком как с обычной колонкой. Например, применять функцию для каждой строчки (то есть для каждого тиббла) с помощью map() и записывать результат в новую колонку с помощью mutate().
heroes %>%
group_by(Gender) %>%
nest() %>%
mutate(dim = map(data, dim))В конце концов нам нужно “разжать” сжатую колонку-список. Сделать это можно с помощью unnest(), выбрав с помощью tidyselect нужные колонки.
heroes %>%
group_by(Gender) %>%
nest() %>%
mutate(dim = map(data, dim)) %>%
unnest(dim)Разжатая колонка обычно больше сжатой, поэтому разжатие привело к удлинению тиббла. Вместо удлинения тиббла, его можно расширить с помощью unnest_wider().
heroes %>%
group_by(Gender) %>%
nest() %>%
mutate(dim = map(data, dim)) %>%
unnest_wider(dim, names_sep = "_") Нестинг - это мощный инструмент tidyverse, хотя во многих случаях можно обойтись и без него. Наиболее эффективна эта конструкция именно в тех ситуациях, где вы делаете операции над целыми тибблами. Поэтому наибольшее распространение нестинг получил в смычке с пакетом broom для расчета множественных статистических моделей.
Функция
across()появилась в пакетеdplyrотносительно недавно, до этого для работы с множественными колонками в tidyverse использовались многочисленные функции*_at(),*_if(),*_all(), например,summarise_at(),summarise_if(),summarize_all(). Эти функции до сих пор присутствуют вdplyr, но считаются устаревшими. Другая альтернатива - использование пакетаpurrr(9.4) или семейства функцийapply()(@ref(apply_f)).↩︎Если ключи будут неуникальными, то функции
*_join()не будут выдавать ошибку. Вместо этого они добавят в итоговую таблицу все возможные пересечения повторяющихся ключей. С этим нужно быть очень осторожным, поэтому рекомендуется, во-первых, проверять уникальность ключей на входе и, во-вторых, проверять тиббл на выходе. Ну или использовать эту особенность работы функции*_join()себе во благо.↩︎